Biblioteca Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Sanità Pubblica Veterinaria: Numero 100, Febbraio 2017 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#652
Tecniche diagnostiche "non convenzionali" per l'identificazione di dermatofiti in campo umano e veterinario: sequenziamento genico vs MALDI-TOF MS
Unconventional diagnostic techniques for dermatophytes identification in human and veterinary medicine: gene sequencing vs MALDI-TOF MS
Deborah Cruciani1, Manuela Papini2, Francesco Agnetti1, Simone Caruso5, Laura Madeo1, Paola Papa1, Cristina Pesca1, Elisa Sgariglia1, Daniele Paoloni4, Silvia Crotti1, Lucia Pitzurra3
1 Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
2 Dipartimento di Scienze Chirurgiche e Biomediche , Università di Perugia, Clinica Dermatologica di Terni
3 Dipartimento di Medicina Sperimentale, Sezione di Microbiologia, Facoltà di Medicina e Chirurgia, Perugia
4 Istituto Oikos s.r.l., Milano
5 Laurea Magistrale in Ecobiologia, Filadelfia (VV)
Abstract. The growing attention to fungal infections, observed both in human and veterinary cases, resulted in an innovative diagnostic approach, based on molecular biology and mass spectrometry techniques to overcome limits of traditional methods. In particular, in this study we compare two fungal identification systems: gene sequencing and MALDI-TOF MS
Riassunto.La crescente attenzione verso le micosi, riscontrata sia in campo umano che veterinario, ha determinato un approccio diagnostico innovativo, basato su tecniche di biologia molecolare e spettrometria di massa, che superano i limiti del tradizionale esame colturale. In particolare, in questo studio si confrontano due sistemi di identificazione fungina: sequenziamento genico e MALDI-TOF MS
Introduzione
I dermatofiti sono funghi cheratinofili capaci di invadere i tessuti cheratinizzati dei mammiferi, causando spesso infezioni contagiose (Weitzman e Summerbell 1995) di tipo superficiale, note come dermatofitosi o tigne o ringworm (Graser et al., 1999). L'attenzione verso le dermatofitosi è cresciuta notevolmente negli ultimi anni, non solo in campo umano, per le opportune valutazioni cliniche e scelte terapeutiche, ma anche in campo veterinario, poiché tutti i dermatofiti hanno un potenziale ruolo zoonosico (Buchanan e Ledeboer 2013; Freydiere et al., 2001; Moretti et al., 2013).
Ciò ha spinto a condurre un'indagine più accurata, applicando a campioni veterinari ed umani tecniche diagnostiche innovative.
La diagnosi di dermatofitosi è stata a lungo basata esclusivamente sull' esame colturale, che implica la valutazione di tutti gli aspetti macroscopici e microscopici del fungo in esame. Questo tipo di approccio, considerato "tradizionale", può avere un esito non esaustivo, soprattutto se l'obiettivo è quello di condurre un'indagine epidemiologica accompagnata da una diagnosi di specie. Molto spesso, infatti, l'esame colturale consente l'identificazione del genere, ma non della specie a cui appartiene il patogeno, che invece fornirebbe informazioni più dettagliate.
Altra forte limitazione dell'esame colturale è la possibilità di contaminazione da parte di muffe non dermatofitiche, che può rendere difficoltosa l'interpretazione del patogeno (Agnetti et al., 2004). Inoltre, questa procedura è di tipo "time-consuming", poiché richiede fino a 15 giorni di incubazione, causando ritardi nella prescrizione dell'eventuale trattamento terapeutico [Weitzman e Summerbell 1995).
I recenti sviluppi nel campo della diagnostica molecolare e della spettrometria di massa hanno consentito di approcciare alla diagnosi di dermatofitosi con metodi più innovativi (Buchanan e Ledeboer 2013; Cafarchia et al., 2009, 2013), superando le suddette limitazioni.
In questo studio, sono stati confrontati i risultati ottenuti con le metodiche del sequenziamento genico e MALDI-TOF MS (Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry) su dermatofiti isolati da uomo e animali.
Materiali e metodi
Sono stati analizzati 21 campioni totali, di cui 9 di origine umana e 12 animale. I campioni umani, rappresentati da unghie, sono stati prelevati dal piede di pazienti diabetici seguiti presso l'Azienda Ospedaliera di Terni e aderenti ad uno studio sistematico epidemiologico e clinico sulla presenza di Tinea pedis e onicomicosi come complicanze del diabete (progetto ospedaliero iniziato nel gennaio del 2015). Peli di origine animale sono stati invece prelevati da cani e gatti affetti da dermatopatie e da scoiattoli sottoposti a screening micologici nell'ambito del Progetto LIFE U-SAVEREDS, sulla salvaguardia dello scoiattolo rosso (LIFE + 2013).
I campioni sono stati seminati su Dermasel Agar e incubati a 25°±1°C in aerobiosi fino a 15 giorni. La crescita fungina è stata monitorata ogni 2-3 giorni, fino al termine del periodo di incubazione. Ciascuna colonia, una volta in purezza, è stata identificata con le metodiche tradizionali, osservando quindi le caratteristiche macroscopiche e microscopiche; successivamente la stessa è stata sottoposta ad indagini molecolari e di spettrometria.
Per le analisi molecolari si è provveduto a prelevare una porzione di colonia pari a circa ½ cm2 e ad estrarre il DNA tramite bollitura, in 200 ?L di soluzione fisiologica. Il DNA è stato quindi amplificato tramite Hemi-Nested PCR utilizzando i seguenti primer:
- DMTF18SF1 5'-CCAGGGAGGTTGGAAACGACCG-3'
- DMTF28SR1 5'-CTACAAATTACAACTCGGACCC-3'
- DMTFITS1R 5'- CCGGAACCAAGAGATCCGTTGTTG-3'
La miscela di reazione e le condizioni di amplificazione sono riportate in bibliografia (Cafarchia et al., 2009, 2013). I prodotti di PCR sono stati purificati tramite QIAquick PCR Purification Kit (Qiagen®), sottoposti a reazione di sequenza con BigDye Terminator v1.1 Cycle Sequencing Kit e analisi in sequenziatore automatico ABI PRISM 310 Genetic Analyzer (Applied Biosystems).
I ferogrammi ottenuti sono stati elaborati con il software BioEdit Sequence Alignment Editor v 7.0.9.0 per generare la sequenza consenso, infine allineata con il database presente in GenBank.
In parallelo, le medesime colonie sono state analizzate mediante tecnologia MALDI-TOF MS, utilizzando il MicroFlex LT system (Bruker®).
I campioni sono stati preparati secondo il protocollo di Saffert et al. (Saffert et al., 2011), quindi 1 µL di estratto proteico è stato depositato sulla piastra e, dopo completo assorbimento, è stato addizionato 1µL di matrice per la successiva analisi.
Per la validazione dei due metodi, ad ogni sessione analitica è stato aggiunto un ceppo di riferimento di Microsporum canis (ATCC 6299).
Risultati
I valori relativi al ceppo ATCC sono risultati soddisfacenti per entrambe le metodiche, validando quindi i risultati ottenuti, riportati in Tabella 1.
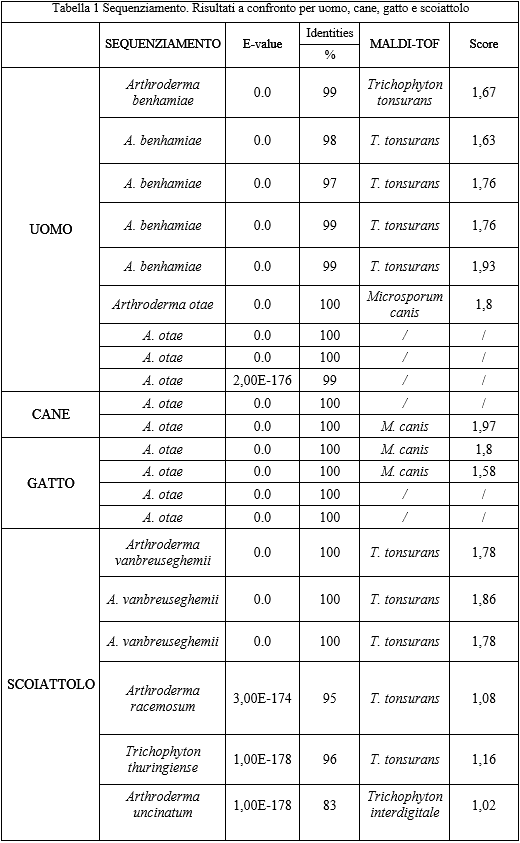
Table 1. Sequencing. Comparative results for man, dog, cat and squirrel
Dei 21 campioni analizzati in doppio, è stato possibile confrontarne solo 15, poichè in 6 casi (28,6%) la tecnologia MALDI-TOF non è stata in grado di identificare alcuna specie fungina.
Il sequenziamento eseguito sui 21 ceppi fungini ha confermato nel 100% dei casi l'esito dell'esame colturale tradizionale. Inoltre, i dati ottenuti sono considerati ampiamente attendibili in base a specifici parametri, quali percentuale d'identità ed E-value. Ciò vale in particolar modo per i dermatofiti zoofili più diffusi in natura, come Arthroderma otae (forma sessuata di M. canis) e A. vambreuseghemii/A. benhamiae (forma sessuata di Trichophyton mentagrophytes); per i dermatofiti geofili, quali T. thuringiense, A. uncinatum (forma sessuata di T. ajelloi) e A. racemosum (forma sessuata di M. cookei) isolati dallo scoiattolo grigio americano, le percentuali di identità sono più basse ma comunque sempre soddisfacenti.
I risultati ottenuti con il MALDI-TOF MS sono confrontati con quelli del sequenziamento in Tabella 1, dove si osserva per lo più concordanza nel riconoscimento del genere, non della specie (10 isolati, 66,7%). Solo in un caso (6,7%) non c'è stata corrispondenza nemmeno per il genere (M. cookei vs T. tonsurans).
Il dermatofita zoofilo M. canis, invece, è stato correttamente identificato con entrambi i metodi diagnostici (4 isolati, 26,7%).
Il parametro che indica l'attendibilità del risultato fornito dal MALDI-TOF MS è lo score. Secondo Saffert R. T. et al.(2011) uno score ≥ 2,000 indica l'identificazione di specie, mentre uno score compreso tra 1,700 e 1,999 indica l'identificazione di genere; infine uno score < 1,700 viene interpretato come una mancata identificazione. Dai valori riportati in Tabella 1 si nota che questa "ripartizione" non è sempre perfettamente rispettata, soprattutto per quanto riguarda la corrispondenza di specie, per la quale i valori di score tendono comunque a 2.
Discussione
I dati in nostro possesso sono sicuramente esigui per poter essere considerati statisticamente significativi, ma, seppur preliminari consentono già alcune osservazioni.
In primo luogo, si nota che grazie al sequenziamento è sempre possibile l'attribuzione di specie, anche nei casi in cui l'approccio tradizionale non sia risolutivo; inoltre, si può desumere che la banca dati relativa alle specie fungine utilizzata dallo strumento MALDI-TOF MS andrebbe sicuramente "popolata" in modo tale da fornire dati utili a scopo diagnostico; infine, c'è da considerare che la matrice di partenza utilizzata, rappresentata dalla colonia fungina in terreno colturale solido, potrebbe non essere quella più idonea alla metodica di spettrometria di massa riportata in questo lavoro.
Si rende quindi necessario indagare un numero maggiore di ceppi fungini a partire da terreni di coltura diversi o effettuare l'estrazione di proteine sulle diverse strutture del micete, analizzando separatamente spore ed ife.
I dati pubblicati in questo lavoro riguardanti la popolazione di scoiattolo grigio
sono stati raccolti durante il Progetto LIFE13 BIO/IT/000204 "Management of grey squirrel in Umbria:
conservation of red squirrel and preventing loss of biodiversity in Apennines" (LIFE U-SAVEREDS).
Bibliografia
Agnetti F., Boncio L., Panzieri C. (2004). Diagnosi di laboratorio delle Dermatofitosi. Sanità Pubblica Veterinaria, n. 23, Gennaio Febbraio. http://spvet.it/arretrati/numero_23/dermatof.html
Buchanan B. W., Ledeboer N. A. (2013). Advances in identification of clinical yeast isolates by use of Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass spectrometry. Journal of Clinical Microbiology, 51(5): 1359-1366.
Cafarchia C., Gasser R. B., Figueredo L. A., Weigl S., Danesi P., Capelli G., Otranto D. (2013). An improved molecular diagnostic assay for canine and feline dermatophytosis. Medical Mycology 51, 136-143
Cafarchia C., Otranto D., Weigl S., Campbell B. E., Parisi A., Cantacessi C., Mancianti F., Danesi P., Gasser R. B. (2009). Molecular carachterization of selected dermatophytes and their identification by electrophoretic mutation scanning. Electrophoresis , 30, 3555-3564.
Freydiere A. M., Guinet R., Boiron P. (2001). Yeast identification in the clinical microbiology laboratory: phenotypical methods. Medical Mycology, 39: 9-33.
Graser Y., El Fari M., Vilgalys R., Kuijpers A.F. A, De Hoog G. S., Presber W., Tietz H. J. (1999). Phylogeny and taxonomy of the family Arthrodermataceae (dermatophytes) using sequence analysis of the ribosomal ITS region. Medical Mycology, 37: 105-114.
Moretti A., Agnetti F., Mancianti F., Nardoni S., Righi C., Moretta I., Morganti G., Papini M. (2013). Dermatophytosis in animals: epidemiological, clinical and zoonotic aspects. Giornale Italiano di Dermatologia e Venereologia, 148:563-572.
Saffert R. T., Cunningham S. A., Ihde S. M., Jobe K. E., Mandrekar J., Patel R. (2011).
Comparison of Bruker biotyper matrix - assisted laser desorption ionization - time of flight mass spectrometer to BD Phoenix automated microbiology system for identification of Gram-negative bacilli. Journal of Clinical Microbiology, 887-892.
Weitzman I., Summerbell R. C. (1995). The dermatophytes. Clinical Microbiology Reviews, 8: 240-259.

Cruciani et al., 2017. Tecniche diagnostiche "non convenzionali" per l'identificazione di dermatofiti in campo umano e veterinario: sequenziamento genico vs MALDI-TOF MS. (SPVet.it 100/2017)



