Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#693
EDITORIALE:
Allergeni vegetali: individuazione e validazione di sistemi efficienti da applicare in fast PCR real-time
Plant allergens: identification and validation of efficient systems to apply in fast real-time PCR
[Ver. 1.0]
Martina Torricelli, Elisa Pierboni, Naceur Haouet, Maria Serena Altissimi, Gloria Raquel Tovo, Sabrina Zampa, Cristina Rondini
Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche "Togo Rosati". Perugia
Abstract. Food allergy has become an important health concern worldwide. The main health risk for sensitized individuals is due to the presence of traces of allergens results of an accidental contamination during food processing and so of undeclared allergenic ingredients in label. Therefore, reliable analytical methods are required to detect and identify allergenic ingredients in food products. PCR techniques allowed a specific amplification of allergen sequences. Furthermore, some processing methods may induce the protein denaturation, the fragmentation and/or the degradation of genomic DNA, so it is necessary to perform some studies in order to analyze the effect of processing on the detection of different targets. In this work, we want to present real-time PCR validated methods to apply for the detection of some plant allergens also in processed food products
Riassunto. Attualmente, le allergie alimentari rappresentano una preoccupazione sanitaria globale. Il rischio principale per gli individui suscettibili è dovuto alla presenza di tracce di allergeni, risultato di contaminazioni accidentali durante il processo di trasformazione alimentare e quindi di ingredienti allergenici non dichiarati in etichetta. Per questo, sono richiesti metodi analitici affidabili per rilevare e identificare gli ingredienti allergenici nei prodotti alimentari. Le tecniche di PCR permettono un'amplificazione specifica delle sequenze e dei target allergenici. Inoltre, alcuni metodi di lavorazione potrebbero indurre la denaturazione proteica, la frammentazione e/o la degradazione del DNA genomico, quindi sono necessari degli studi finalizzati ad analizzare l'effetto delle trasformazioni dell'alimento sulla rilevazione dei differenti target. Lo scopo di questo lavoro è di presentare metodi in PCR real-time validati, da applicare per la rilevazione di alcuni allergeni vegetali anche in prodotti alimentari trasformati
Introduzione
L'Allergia Alimentare (AA) è una manifestazione mediata da reazioni immunologiche specifiche verso determinanti allergenici contenuti negli alimenti, in cui alcune molecole (principalmente proteine), sono in grado di causare reazioni immediate o ritardate di diversa gravità per gli individui suscettibili (Allergie alimentari e sicurezza del consumatore - Documento di indirizzo e stato dell'arte Italia 2018, Ministero della Salute). Tra le ipersensibilità alimentari, la sintomatologia e il quadro clinico possono variare infatti ampiamente, dal leggero malessere gastrointestinale, rush cutanei, crisi respiratorie, caratterizzate da asma, fino allo shock anafilattico, dipendente dalla dose, diversa sensibilità individuale, stato di salute, circostanza e allergene coinvolto.
Le allergie alimentari sono considerate un fenomeno in forte crescita, anche se non devono essere confuse con le reazioni tossiche, con le intolleranze farmacologiche (per es. da istamina…) e con intolleranze da difetti enzimatici o intolleranze alimentari vere, come quella verso il lattosio (data dalla mancanza dell'enzima lattasi deputato alla digestione dello zucchero contenuto nel latte, ovvero il lattosio, scisso in glucosio e galattosio) (Taylor et al., 2004). Occupano un ruolo rilevante altre reazioni immunomediate, quale in particolare la celiachia che sta interessando sempre più individui di differenti fasce di età (Muraro et al. 2014).
In realtà le allergie alimentari, associate ai Paesi industrializzati, hanno insorgenza in età pediatrica e spesso regressione in fase di crescita, oppure manifestazione in età adulta con persistenza per tutta la vita: si stima che nei paesi sviluppati l'AA interessi dal 5% al 10% dei soggetti in età pediatrica ed una percentuale minore di soggetti in età adulta (Koplin et al., 2018). La prevalenza dell'AA presenta una differente distribuzione geografica determinata da differenti abitudini dietetiche e stili di vita, dalle modalità prevalenti di cottura dei cibi, dalle modalità e tempi dello svezzamento nelle diverse popolazioni, dall'esposizione agli aeroallergeni, importante nel determinare i profili di sensibilizzazione nelle varie regioni, ed infine da fattori genetici (Bartra et al., 2016).
Il Regolamento dell'Unione Europea 1169/2011 stabilisce una lista di 14 ingredienti responsabili di allergie e/o intolleranze che devono essere obbligatoriamente dichiarati in etichetta e che comprendono: cereali contenenti glutine, pesci, crostacei, molluschi, latte, uova, senape, sesamo, sedano, lupino, soia, arachide, frutta a guscio (mandorle, nocciole, noci, noci di acagiù, noci pecan, noci del Brasile, pistacchi, noci macadamia o noci del Queensland) e solfiti.
Per quanto riguarda l'etichettatura dei prodotti alimentari, l'applicazione del Parlamento Europeo e del Consiglio ha sancito l'obbligo di evidenziare, nella lista degli ingredienti, gli allergeni presenti nei prodotti alimentari. Inoltre, ai sensi dell'articolo 44 della legislazione vigente, si rende obbligatorio riportare in modo evidenziato la presenza di sostanze o prodotti che provocano allergie o intolleranze anche per gli alimenti non pre-imballati, compresi quelli preparati e forniti da aziende di ristorazione (ristoranti, mense, mense scolastiche…) o imballati sui luoghi di vendita su richiesta del consumatore o per la vendita diretta.
Il paziente allergico è in grado di individuare facilmente e quindi non consumare un determinato alimento, quando la sostanza allergenica costituisce uno degli ingredienti di quel prodotto alimentare ed è riportato nella denominazione di vendita (latte, uova, ecc.). Un aspetto più delicato che richiede un'attenta lettura dell'etichetta riguarda invece la presenza non esplicita e inattesa dei cosiddetti "allergeni nascosti o occulti" presenti in traccia, risultato di fenomeni di cross-contaminazione accidentale lungo le diverse fasi della catena produttiva e della filiera alimentare. Per cercare di gestire tali criticità, l'industria alimentare ha optato per l'adozione di una etichettatura precauzionale volontaria o PAL ("Precautionary Allergen Labelling") prevista e argomentata nell'articolo 36 del Regolamento (UE) n. 1169/2011.
Nonostante ciò, le segnalazioni dovuti alla mancata indicazione in etichetta degli allergeni negli alimenti è in aumento, come si può vedere dalle notifiche RASFF - Rapid Alert System for Food and Feed (Padua et al., 2019).
Con l'etichettatura "precauzionale" il carico della valutazione del rischio sembra quasi essere stato ribaltato sul consumatore, creando frustrazione e insicurezza nella possibilità di acquistare e consumare alimenti sicuri.
Infine, è necessario porre attenzione su un altro aspetto critico che riguarda i trattamenti tecnologici a cui sono sottoposti gli alimenti, che talvolta possono modificare la struttura proteica, influenzando il potenziale allergenico. Per alcuni allergeni (es. arachide) le pratiche di trasformazione industriale (torrefazione o frittura) possono addirittura comportare l'aumento del potenziale allergenico, apportando modifiche che comportano una maggior esposizione dei determinanti antigenici (epitopi) (van Hengel, 2007).
Sulla base di ciò, uno dei modi per poter ovviare a tali problemi tutelando quindi il più possibile la salute dei consumatori allergici è individuare ed impiegare degli approcci analitici validi per l'identificazione di un allergene.
Le tecniche per la rilevazione di potenziali allergeni nei prodotti alimentari sono da considerarsi di due tipologie: approccio diretto (1. identificazione della proteina allergizzante mediante tecniche immunoenzimatiche, quali l'ELISA o il Lateral Flow Device-LFD, 2. identificazione dei peptidi marker mediante metodologia basata sulla spettrometria di massa) e indiretto (identificazione della sua presenza mediante analisi del DNA). Ognuna di queste tecniche presenta vantaggi e svantaggi in termini di resa di estrazione, rapidità d'analisi, specificità di reazione e costi, ma attualmente per l'industria alimentare e i laboratori deputati al controllo ufficiale degli alimenti la tecnica ELISA è il metodo d'elezione più comunemente impiegato nell'analisi di routine.
D'altra parte, la tecnica di amplificazione del DNA genomico totale di un alimento attraverso la reazione a catena della polimerasi (PCR) permette di ottenere marcatori molecolari specifici funzionali non all'identificazione "diretta" dell'allergene, ma piuttosto all'identificazione dei geni che codificano per quel particolare allergene (o di altri geni marker). L'amplificazione di geni diversi codificanti diversi allergeni caratteristici di uno stesso organismo (ad esempio. Cor a 1, Cor a 8, Cor a 14 per nocciola) può condurre a rese di amplificazione diverse, rendendo quindi fondamentale la fase di disegno sperimentale e la selezione delle sequenze marker (D'Andrea et al., 2011).
Di recente, è stata introdotta la tecnica della PCR digitale (Huggett et al., 2015) il cui concetto base è quello di distribuire il DNA genomico presente nel campione in numerose frazioni (diluendo quindi fino ad 1-2 molecole di DNA, potenzialmente) in celle o micro-gocce (droplets) di dimensioni micrometriche, e i cui dati ottenuti vengono poi normalizzati mediante la Legge di Poisson. Un recente lavoro ha visto l'applicazione della droplet digital PCR per la rilevazione e quantificazione degli allergeni arachidi e soia, portando a risultati paragonabili se non più soddisfacenti rispetto alla PCR real-time (Pierboni et al., 2018).
Il Progetto di Ricerca Corrente
In base allo stato dell'arte illustrato, l'obiettivo della Ricerca Corrente 2017 finanziata dal Ministero della Salute, Progetto IZSUM 07/17 RC, era quello di indagare sulle possibili cause e sull'impatto che ha la trasformazione degli alimenti (dalla materia prima al prodotto finito) sul potere antigenico della proteina responsabile, partendo da un'indagine epidemiologica medica delle allergie più diffuse nel territorio umbro-marchigiano. Il potere antigenico stesso viene determinato attraverso l'analisi qualitativa e quantitativa, effettuata mediante metodiche ELISA, PCR real-time e spettrometria di massa a cromatografia liquida, LC-MS, messe quindi a confronto. In particolare l'obiettivo primario della nostra unità operativa è finalizzato a trovare, laddove già esistenti in letteratura, o a costruire, laddove carenti o poco soddisfacenti, sistemi efficienti da validare ed idonei all'analisi e alla rilevazione dei suddetti target allergenici in fast PCR real-time.
L'impiego della PCR real-time ha permesso di investigare alcuni allergeni vegetali riconosciuti dal Regolamento UE 1169/2011 (senape, sedano, nocciola, sesamo, lupino, anacardo, mandorla, pistacchio, noce, noce pecan, noce del Brasile, noce macadamia), in aggiunta a soia e arachide già investigati e validati in un precedente progetto di Ricerca Corrente IZSUM 07/14 (Pierboni et al., 2018). A seguito di un'estrazione del DNA mediante una metodica già validata, è stata eseguita una valutazione quali-quantitativa del DNA tramite fotometro/fluorimetro e test d'inibizione in PCR real-time mediante chimica TaqMan. Per i nuovi target da analizzare, il laboratorio, dopo un'accurata ricerca bibliografica, ha eseguito una valutazione preliminare dei metodi esistenti e la verifica sperimentale di quelli più idonei.
La valutazione dei metodi molecolari si è basata sulla selezione della sequenza nucleotidica ed eventuale disegno di primer e sonde specifici. Per tutti i sistemi considerati è stata eseguita l'ottimizzazione delle concentrazioni oligonucleotidiche e conseguente validazione del metodo considerando la verifica del LOD (Limit Of Detection), della specificità e della robustezza (MPR, 2015).
Per riprodurre condizioni di campioni reali possibilmente reperibili in commercio (sottoposti a trattamenti termici e non) e per valutare il grado di termo-stabilità degli allergeni, anche rispetto al contenuto proteico, sono stati inoltre preparati dei campioni contaminati. Questi campioni o incurred samples, ovvero delle torte sia al cioccolato sia agli agrumi, sono stati ottenuti addizionando al preparato iniziale, 5 gr di soia (0.8%), 5 gr di arachide (0.8%) e 10 g di nocciola sotto forma di crema spalmabile "Nutella" (0.2%), cotte per 60' a differenti temperature: 120°C, 140°C, 165° C (temperatura consigliata nei preparati acquistati) e 190°C.
Nell'ambito del progetto, il lavoro finora svolto dall'unità operativa (Laboratorio OGM e Igiene dell'Ambiente), riguarda principalmente la messa a punto e validazione dei metodi in fast PCR real-time, descritta di seguito.
Materiali e Metodi
Estrazione del DNA
- Le matrici allergeniche oggetto dello studio condotto sino ad oggi sono:
- senape (Sinapis alba), mustard (Family: Brassicaceae o Crucifere)
- sedano (Apium graveolens), celery (Family: Apiaceae)
- nocciola (Corylus avellana), hazelnut (Family: Betulaceae)
- anacardo (Anacardium occidentale), cashew nut (Family: Anacardiaceae)
- sesamo (Sesamum indicum), sesame (Family: Pedaliaceae)
- pistacchio (Pistacia vera), pistachio (Family: Anacardiaceae)
Le analisi molecolari dei target possono essere condotte previa un'efficiente estrazione del DNA in termini quantitativi e qualitativi. Dopo un'opportuna macinazione che ha previsto l'utilizzo di ghiaccio secco in particolare per la frutta a guscio e azoto liquido in caso di foglie e per taluni semi, le matrici d'interesse, incluse quelle impiegate per il test della specificità, sono state estratte usando un metodo individuato e validato per i target arachide e soia nel precedente progetto di ricerca (IZSUM 07/14 RC) e pubblicato in Pierboni et al. 2018: esso consiste nell'impiego del buffer di estrazione CTAB (UNI EN ISO 21571:2013), combinato ad un kit di purificazione del DNA, il NucleoSpin gDNA Clean-up, Macherey Nagel®, aggiungendo una fase di concentrazione, che prevede la riunificazione di due estratti di un campione in un'unica colonnina di purificazione in silica (SPE, solid phase extraction). Per le matrici arachide e anacardo, è stata seguita la stessa procedura senza però la fase aggiuntiva, per evitare una maggiore concentrazione di inibitori. Per le mandorle e le noci si sono ottenuti buoni risultati quantitativi e qualitativi, concentrando o meno il DNA.
Il dosaggio dei DNA estratti è stato eseguito in fluorimetria, mediante lo strumento BioSpectrometer (Eppendorf) e l'impiego del Qubit dsDNA BR Assay Kit (Thermo Fisher Scientific).
Tutti i DNA estratti sono stati inoltre amplificati in duplice copia nel test d'inibizione in fast PCR real-time (MPR, 2015) per il gene universale pianta generico actina (Torricelli et al. 2016), testando sia la concentrazione di DNA tal quale sia la sua diluzione 1:4, per valutare l'eventuale presenza di inibitori e quindi la purezza del DNA.
Fast Real-time PCR
I sistemi di PCR adottati sono illustrati nella Tabella 1 (Table 1). Per gli allergeni senape, sedano e nocciola, sono stati presi in considerazione sistemi validati e normati UNI CEN/TS ("European Committee for standardization"), rispettivamente"MADS-D protein", "mannitol-dehydrogenase gene, MDH", "Pathogenesis-related protein, PR-10, Bet v1-family member" CorA-1, comunque testati nuovamente per valutare il grado di adattabilità alle diverse variabili intra-laboratorio (metodo di estrazione, reagenti utilizzati, in particolare relativamente all'impiego della TaqManTM Fast Universal PCR Master Mix-Applied Biosystem by Thermo Fisher Scientific) e quindi alle condizioni di corsa, in modalità fast che permette di ridurre il tempo di reazione (a circa 40 minuti), mantenendo l'efficienza di amplificazione. Per quanto riguarda l'allergene anacardo, dopo un accurato studio bibliografico è stato scelto un sistema genico già pubblicato (Ehlert et al. 2008) ovvero il "2S Albumin, Ana o 3" poi testato e sottoposto a validazione in-house.
Infine per gli allergeni sesamo e pistacchio, essendo i dati presenti in letteratura carenti o poco soddisfacenti, sono stati costruiti ex novo set di oligonucleotidi (primer/sonda) sul tratto genico codificante per la famiglia proteica della 2S Albumina, mediante l'impiego del software Primer Express v 3.0. Per il pistacchio sono stati scelti e messi a confronto due sistemi oligonucleotidici costruiti in regioni differenti dello stesso gene, un sistema con una sonda marcata in BHQ1, un altro con probe marcata in MGB.
I metodi selezionati sono stati testati in fast PCR real-time per l'ottimizzazione delle concentrazioni di primer e sonde. Il test ha incluso la verifica di tre diverse concentrazioni di primer forward e reverse, combinate tra loro per un totale di 9 combinazioni per ogni sistema di PCR, testate ognuna in quadruplice copia. Per ogni sistema, una volta scelta la migliore concentrazione di primer forward e reverse si è proceduto alla selezione della migliore concentrazione di sonda, tra quattro testate in quadruplice copia. I risultati relativi all'ottimizzazione di ogni sistema di PCR sono illustrati nella Tabella 2 (Table 2).
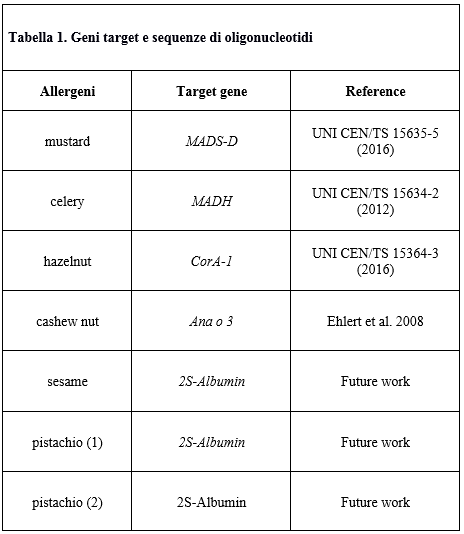
Table 1. Target gene and oligonucleotide sequences
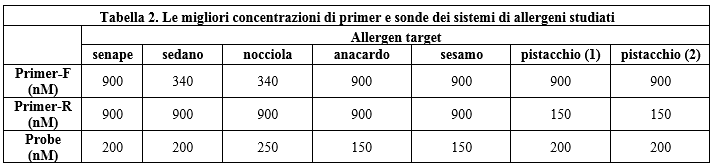
Table 2. The best primer and probe concentrations of the investigated allergen systems
Specificità
Per tutti i metodi in PCR, a monte delle prove sperimentali, sono stati effettuati degli studi in silico, in particolare per valutare la specificità verso le altre specie, sub-specie, generi e/o famiglie, mediante i software BLAST (NCBI) e/o Mega6Alignement. Per quanto riguarda la valutazione di tale parametro, sono state inoltre scelte le specie filogeneticamente e/o botanicamente più vicine e quindi correlate agli allergeni in esame. In totale, sono stati estratti DNA da 57 prodotti diversi costituiti da spezie, frutta, frutta secca, cereali, legumi, tuberi, verdura, specie animali. A seguito di una verifica qualitativa e quantitativa, i DNA sono stati diluiti, per portarli allo stesso ciclo di amplificazione Cq (quantification cycle) per il gene universale actina, al fine di testare la stessa quantità di ogni prodotto con ogni sistema di PCR.
LOD
Al fine di valutare il LOD, ogni sistema oggetto di studio (senape, sedano, nocciola, anacardo, sesamo e pistacchio) è stato testato utilizzando 9 concentrazioni di DNA in ng e/o hge decrescenti, ciascuna testata in 10 replicati. L'ultima diluizione in cui si sono ottenute il 100% di amplificazioni, definita come LOD10, è stata testata in 60 replicati (LOD95), con verifica della diluizione a monte e/o a valle, al fine di scegliere quella con almeno 59 amplificati su 60. E' stato quindi individuato il metodo più sensibile per ciascuna delle matrici, da utilizzare nell'identificazione degli allergeni.
Robustezza
La robustezza per tutti i sistemi oggetto di studio è stata verificata applicando delle piccole variazioni deliberate alla concentrazione di master mix, dei primer e della sonda, al volume di reazione e alla temperatura di annealing, in base a quanto stabilito in Pierboni et al. 2016.
Conclusioni e prospettive future
I metodi biomolecolari individuati sono risultati rapidi, specifici, sensibili, robusti ed affidabili per gli allergeni senape, sedano, nocciola, anacardo, sesamo e pistacchio, soddisfacendo pienamente i parametri di validazione e permettendo quindi di investigare gli allergeni vegetali riconosciuti come tali e inclusi nel Regolamento europeo 1169/2011, a tutela del consumatore. Come già accennato in precedenza, uno degli aspetti più delicati e più critici riguarda la presenza negli alimenti di allergeni in traccia o dei cosiddetti "allergeni nascosti", dovuti a cross-contaminazioni o all'introduzione involontaria lungo le diverse fasi della catena produttiva e della filiera alimentare: dai dati ottenuti dalla validazione dei sistemi di PCR, ne deriva che la sensibilità e la specificità dei metodi bio-molecolari potrebbero contribuire ad allertare sulla la presenza di ingredienti allergenici non etichettati (indipendentemente dalla presenza e dalla rilevazione della frazione proteica), tutelando maggiormente il consumatore e la sua salute. Dai risultati ottenuti nella prova che ha previsto l'impiego dei campioni reali contaminati (incurred samples) si è evidenziato che l'aumento della temperatura, non pregiudica una corretta rilevazione del target allergenico specifico mediante PCR real-time. Sulla base di ciò, l'impiego anche combinato delle tecnologie analitiche ad oggi disponibili, incluse quelle molecolari di ultima generazione (PCR real-time e/o digital PCR) potrebbero in una prospettiva futura superare alcuni limiti tuttora presenti e ottenere un miglioramento in termini di analisi e di sicurezza alimentare.
Bibliografia
Allergie alimentari e sicurezza del consumatore - Documento di indirizzo e stato dell'arte Italia 2018, Ministero della Salute.
Bartra J, Garcia-Moral A, Enrique E. (2016). Geographical differences in food allergy. Bundesgesundheitsblatt Gesundheitsforschung Gesundheitsschutz 59:755-63.
D'Andrea M., Coïsson J.D., Locatelli M., et al. (2011).Validating allergen coding genes (Cor a 1, Cor a 8, Cor a 14) as target sequences for hazelnut detection via Real-Time PCR. Food Chemistry 124:1164-1171.
Ehlert A., Hupfer C., Demmel A., Engel K.H, Busch U. (2008). Detection of Cashew Nut in Foods by a Specific Real-time PCR Method. Food Analytical Methods Issue 2 pp 136-143.
Huggett J.F., Cowen S, Foy C.A. (2015). Considerations for digital PCR as an accurate molecular diagnostic tool. Clinical Chemistry 61:79-88. ISO 21571:2005/Amd.1:2013 "Foodstuffs - Methods of analysis for the detection of genetically modified organisms and derived products - Nucleic acid extraction".
Koplin J.J, Peters R.L, Allen K.L. (2018). Prevention of Food Allergies. Immunology and Allergy Clinics of North America 38:1-11.
MPR 2015. Definition of Minimum Performance Requirements for Analytical Methods of GMO Testing. 20 October 2015.
Muraro A., Worm R.G., Bilò M.B. et al. (2014). Anaphylaxis: guidelines from the European Academy of Allergy and Clinical Immunology. Allergy 69:1026-45.
Pádua I, Moreira A, Moreira P, Melo de Vasconcelos F, Barrosa R, (2019). Impact of the regulation (EU) 1169/2011: Allergen-related recalls in the rapid alert system for food and feed (RASFF) portal. Food control 98:389-398.
Pierboni E., Curcio L., Tovo G.R., Torricelli M., Rondini C. (2016). Evaluation of Systems for Nopaline Synthase Terminator in Fast and Standard Real-Time PCR to Screen Genetically Modified Organisms. Food Analytical Methods, Volume 9, Issue 4, pp 1009-1019, doi: 10.1007/s12161-015-0283-7.
Pierboni E., Rondini C., Torricelli M., Ciccone L., Tovo G.R., Mercuri M.L., Altissimi S., Haouet N. (2018). Digital PCR for analysis of peanut and soybean allergens in foods. Food Control 92:128-136.
Regolamento (UE) N. 1169/2011 DEL PARLAMENTO EUROPEO E DEL CONSIGLIO del 25 ottobre 2011 relativo alla fornitura di informazioni sugli alimenti ai consumatori, che modifica i regolamenti (CE) n. 1924/2006 e (CE) n. 1925/2006 del Parlamento europeo e del Consiglio e abroga la direttiva 87/250/CEE della Commissione, la direttiva 90/496/CEE del Consiglio, la direttiva 1999/10/CE della Commissione, la direttiva 2000/13/CE del Parlamento europeo e del Consiglio, le direttive 2002/67/CE e 2008/5/CE della Commissione e il regolamento (CE) n. 608/2004 della Commissione.
Taylor S.L., Hefle .SL., Bindsley-Jensen C., et al. (2004). A consensus protocol for the determination of the threshold doses for allergenic foods: how much is too much? Clinical and Experimental Allergy 34:689-695.
Torricelli M., Pierboni E., Tovo G. R., Rondini C. (2016). In-house validation of a DNA extraction protocol from honey and bee pollen and analysis in Fast real-time PCR of commercial honey samples using a knowledge-based approach. Food Analytical Methods doi: 10.1007/s12161-016-0539-x.
UNI CEN/TS 15634-2:2012. Foodstuffs - Detection of food allergens by molecular biological methods - Part 2: Celery (Apium graveolens) - Qualitative determination of a specific DNA sequence in cooked sausages by real-time PCR.
UNI CEN/TS 15634-5:2016. Foodstuffs - Detection of food allergens by molecular biological methods - Part 5: Mustard (Sinapis alba) and soya (Glycine max) - Qualitative detection of a specific DNA sequence in cooked sausages by real-time PCR.
UNI CEN/TS 15634-3:2016. Foodstuffs - Detection of food allergens by molecular biological methods - Part 3: Hazelnut (Corylus avellana) - Qualitative detection of a specific DNA sequence in chocolate by real-time PCR.
Van Hengel A. J. (2007). Food allergen detection methods and the challenge to protect food-allergic consumers. Anaytical Bioanalytical Chemistry 389:111-118.
D'Andrea M., Coïsson J.D., Locatelli M., et al. (2011).Validating allergen coding genes (Cor a 1, Cor a 8, Cor a 14) as target sequences for hazelnut detection via Real-Time PCR. Food Chemistry 124:1164-1171.
Ehlert A., Hupfer C., Demmel A., Engel K.H, Busch U. (2008). Detection of Cashew Nut in Foods by a Specific Real-time PCR Method. Food Analytical Methods Issue 2 pp 136-143.
Huggett J.F., Cowen S, Foy C.A. (2015). Considerations for digital PCR as an accurate molecular diagnostic tool. Clinical Chemistry 61:79-88. ISO 21571:2005/Amd.1:2013 "Foodstuffs - Methods of analysis for the detection of genetically modified organisms and derived products - Nucleic acid extraction".
Koplin J.J, Peters R.L, Allen K.L. (2018). Prevention of Food Allergies. Immunology and Allergy Clinics of North America 38:1-11.
MPR 2015. Definition of Minimum Performance Requirements for Analytical Methods of GMO Testing. 20 October 2015.
Muraro A., Worm R.G., Bilò M.B. et al. (2014). Anaphylaxis: guidelines from the European Academy of Allergy and Clinical Immunology. Allergy 69:1026-45.
Pádua I, Moreira A, Moreira P, Melo de Vasconcelos F, Barrosa R, (2019). Impact of the regulation (EU) 1169/2011: Allergen-related recalls in the rapid alert system for food and feed (RASFF) portal. Food control 98:389-398.
Pierboni E., Curcio L., Tovo G.R., Torricelli M., Rondini C. (2016). Evaluation of Systems for Nopaline Synthase Terminator in Fast and Standard Real-Time PCR to Screen Genetically Modified Organisms. Food Analytical Methods, Volume 9, Issue 4, pp 1009-1019, doi: 10.1007/s12161-015-0283-7.
Pierboni E., Rondini C., Torricelli M., Ciccone L., Tovo G.R., Mercuri M.L., Altissimi S., Haouet N. (2018). Digital PCR for analysis of peanut and soybean allergens in foods. Food Control 92:128-136.
Regolamento (UE) N. 1169/2011 DEL PARLAMENTO EUROPEO E DEL CONSIGLIO del 25 ottobre 2011 relativo alla fornitura di informazioni sugli alimenti ai consumatori, che modifica i regolamenti (CE) n. 1924/2006 e (CE) n. 1925/2006 del Parlamento europeo e del Consiglio e abroga la direttiva 87/250/CEE della Commissione, la direttiva 90/496/CEE del Consiglio, la direttiva 1999/10/CE della Commissione, la direttiva 2000/13/CE del Parlamento europeo e del Consiglio, le direttive 2002/67/CE e 2008/5/CE della Commissione e il regolamento (CE) n. 608/2004 della Commissione.
Taylor S.L., Hefle .SL., Bindsley-Jensen C., et al. (2004). A consensus protocol for the determination of the threshold doses for allergenic foods: how much is too much? Clinical and Experimental Allergy 34:689-695.
Torricelli M., Pierboni E., Tovo G. R., Rondini C. (2016). In-house validation of a DNA extraction protocol from honey and bee pollen and analysis in Fast real-time PCR of commercial honey samples using a knowledge-based approach. Food Analytical Methods doi: 10.1007/s12161-016-0539-x.
UNI CEN/TS 15634-2:2012. Foodstuffs - Detection of food allergens by molecular biological methods - Part 2: Celery (Apium graveolens) - Qualitative determination of a specific DNA sequence in cooked sausages by real-time PCR.
UNI CEN/TS 15634-5:2016. Foodstuffs - Detection of food allergens by molecular biological methods - Part 5: Mustard (Sinapis alba) and soya (Glycine max) - Qualitative detection of a specific DNA sequence in cooked sausages by real-time PCR.
UNI CEN/TS 15634-3:2016. Foodstuffs - Detection of food allergens by molecular biological methods - Part 3: Hazelnut (Corylus avellana) - Qualitative detection of a specific DNA sequence in chocolate by real-time PCR.
Van Hengel A. J. (2007). Food allergen detection methods and the challenge to protect food-allergic consumers. Anaytical Bioanalytical Chemistry 389:111-118.

Torricelli 2019 - Allergeni vegetali: individuazione e validazione di sistemi efficienti da applicare in fast PCR real-time (SPVet.it 113/2019)

Allergeni vegetali: individuazione e validazione di sistemi efficienti da applicare in fast PCR real-time by Torricelli et al., 2019 is licensed under a Creative Commons Attribution 4.0 International License.
Permissions beyond the scope of this license may be available at http://indice.spvet.it/adv.html.