Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#694
Fattori genetici di resistenza/suscettibilità alla Scrapie Classica nella specie caprina
Genetic factors of resistance / susceptibility to Classical Scrapie in caprine species
[Ver. 1.0]
Martina Torricelli(1), Carla Sebastiani(1), Marcella Ciullo(1), Simone Ceccobelli(2),
Barbara Chiappini(3), Gabriele Vaccari(3), Emiliano Lasagna(2), Francesca Maria Sarti(2), Massimo Biagetti(1)
Barbara Chiappini(3), Gabriele Vaccari(3), Emiliano Lasagna(2), Francesca Maria Sarti(2), Massimo Biagetti(1)
1 Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche "Togo Rosati". Perugia
2 Dipartimento di Scienze Agrarie, Alimentari e Ambientali, Università degli Studi di Perugia
3 Dipartimento di Sicurezza Alimentare, Nutrizione e Sanità Pubblica Veterinaria- Istituto Superiore di Sanità. Roma
2 Dipartimento di Scienze Agrarie, Alimentari e Ambientali, Università degli Studi di Perugia
3 Dipartimento di Sicurezza Alimentare, Nutrizione e Sanità Pubblica Veterinaria- Istituto Superiore di Sanità. Roma
Abstract. The present study was aimed at analyzing the sequence of PRNP gene in 10 Italian different goat populations (Camosciata delle Alpi, Saanen, Grigia Ciociara, Bianca Monticellana, Capestrina, Fulva del Lazio, Facciuta della Valnerina, Teramana, Garganica and Grigia Molisana) and in crossbreed goats, bred in central and southern Italy to evaluate the distribution of the K222 allele, which confers resistance to scrapie in goats
Riassunto. Il presente studio ha lo scopo di analizzare la sequenza del gene PRNP in 10 diverse popolazioni caprine (Camosciata delle Alpi, Saanen, Grigia Ciociara, Bianca Monticellana, Capestrina, Fulva del Lazio, Facciuta della Valnerina, Teramana, Garganica and Grigia Molisana) e un gruppo di meticce, allevate nell'Italia centro meridionale, per valutare la distribuzione dell'allele K 222 che conferisce resistenza alla scrapie nelle capre
Introduzione
La scrapie appartiene alla famiglia di malattie neurodegenerative, lentamente progressive e invariabilmente fatali chiamate EST (Encefalopatie Spongiformi Trasmissibili) che possono colpire sia l'uomo che gli animali (Hunter, 1997; Glatzel e Aguzzi, 2001). Le principali EST che colpiscono l'uomo sono: Creutzfeldt-Jakob Desease (CJD), Insonnia Familiare Fatale (FFI), la sindrome di Gerrstmann-Straussler-Scheinker (GSS) e il Kuru (Prusiner, 2013).
Negli animali le principali EST sono: Encefalopatia Spongiforme Bovina (BSE) che colpisce i bovini, Chronic Wasting Disease (CWD) che colpisce i cervidi (CWD), Trasmissibile Mink Encephalopathy (TME) nel visone, Feline Spongiform Encephalopathy (FSE) nei gatti, la Scrapie che colpisce ovini e caprini(Goldman, 2008) e la recentemente descritta malattia da prioni dei dromedari (CPD) (Babelhadj et al., 2018). La scrapie degli ovicaprini viene comunemente descritta come scrapie classica o atipica (NOR 98) (Vaccari et al., 2009); quest'ultima ha una bassa incidenza ed è considerata una forma sporadica. La scrapie è caratterizzata dall'accumulo nel sistema nervoso centrale (SNC), di una proteina patologica prionica (PrPSc), un'isoforma aberrante della proteina prionica cellulare PrPC (codifica ospite) (Prusiner, 1991).
La forma classica è diffusa in tutto il mondo con le eccezioni di Australia e Nuova Zelanda, dove la malattia è stata eradicata e colpisce tutti i piccoli ruminanti. Il periodo di incubazione dipende da diversi fattori quali: la dose infettiva, l'età dell'animale all'infezione, la via di trasmissione e il genotipo dell'ospite che svolge un ruolo cruciale nella modulazione della suscettibilità alla malattia (Hunter, 1997; Agrimi et al., 2003; Baylis e Goldmann, 2004; Aguzzi et al., 2007; Aguilar-Calvo et al., 2014).
La suscettibilità o resistenza alla scrapie negli ovini e nei caprini dipende dal genotipo del gene PrP dell'ospite e dal ceppo infettivo. Sebbene il potenziale zoonotico di alcuni ceppi di Scrapie sia stato suggerito da vari studi (Cassard et al., 2014; Comoy et al., 2015), ad oggi non ci sono prove di rischio per la popolazione umana come riportato dall'Autorità Europea per la Sicurezza Alimentare (EFSA). La proteina prionica PrP (gene PRNP) ovina codifica per 256 aminoacidi, le posizioni degli aminoacidi nella proteina (e dei relativi codoni nel gene) sono numerati da 1 a 256. E' noto che negli ovini alcuni codoni della PrP sono polimorfici, cioè hanno una sostituzione di una singola base del DNA chiamati SNP (SNP=Single Nucleotide Polymorphisms) e in grado di condizionare la suscettibilità/resistenza alla scrapie.
Tali SNP possono essere non conservative (con cambio di amminoacido) o conservative (senza cambio di amminoacido). I polimorfismi più rilevanti sono quelli situati ai codoni 136, 154 e 171 che sono stati associati a resistenza /suscettibilità. In particolare il codone 136 può codificare per alanina (A) o valina (V), il codone 154 per arginina (R) o istidina (H) e il codone 171 per glutammina (Q) arginina (R)) o istidina (H). La combinazione di questi amminoacidi dà cinque diversi alleli (evidenziati in Europa): VRQ, ARQ, AHQ, ARH e ARR. I genotipi VRQ/VRQ, VRQ/ARQ e ARQ/ARQ sono associati ad un'elevata suscettibilità alla scrapie classica, mentre il genotipo ARR/ARR è stato associato al più alto livello di resistenza alla malattia.
Su questa base, secondo la decisione EU 2003/1/EC (EU 2003), è stato avviato un programma di selezione genetica adottato da ciascuno Stato membro nella Comunità europea per aumentare la frequenza dell'allele ARR associato alla resistenza nelle popolazioni ovine. Nei caprini, il PRNP è altamente polimorfico, oggi, nelle razze caprine mondiali sono stati rilevati almeno 47 polimorfismi incluse mutazioni silenti, e una variante PRNP contenente tre ripetizioni di octapeptidi invece delle solite cinque ripetizioni (Vaccari et al., 2009; Fragkiadaki et al., 2011).
Molti studi europei hanno dimostrato che varianti alleliche del gene PRNP possono modulare la resistenza o la suscettibilità alla scrapie classica. Di tutti i polimorfismi, almeno 16 sono mutazioni silenti, mentre le altre causano una sostituzione aminoacidica, e di queste, almeno sette, sono state correlate con una minore suscettibilità alla scrapie classica nella capra.
In particolare, G127S (Goldmann, 2011), I142M (Goldmann et al., 1996; Fast et al., 2017), H143R (Billinis et al., 2002), N146S/D (Papasavva-Stilianou et al. 2007; Papasavva-Stilianou et al. 2011), R154H (Billinis et al., 2002;Papasavva-Stilianou et al.,2007; Barillet, 2009; Vaccari, 2006), R211Q (Barillet, 2009; Srithakumar et al., 2016) e Q222K (Vaccari et al., 2006; Barillet, 2009; Acutis et al., 2006).
Tuttavia, l'istidina al codone 154 (H154) era chiaramente identificata come un fattore di rischio per la scrapie atipica (NOR98) (Colussi et al., 2008). Quindi basato sul "peso delle prove" e sulla "forza di resistenza", l'EFSA ha stilato un ranking di alleli che vanno dal più resistente al meno resistente: K222 > D146 = S146 > Q211 = H154 = M142 > S127 = H143 > wild type. Quindi, secondo le raccomandazioni EFSA, gli alleli K222, insieme al S146 e D146 potrebbero rappresentare i principali SNPs candidati per avere una buona resistenza ai ceppi di scrapie classica circolanti in Europa.
Il peso dell'evidenza scientifica per la resistenza del K222 è maggiore rispetto a quello per S146 e D146 e per l'allele ARR negli ovini. Tuttavia, la resistenza osservata può essere riferita in modo chiaro ai soli ceppi circolanti nella popolazione caprina Europea ed ai ceppi con cui sono state sperimentalmente inoculate(EFSA Journal, 2017); inoltre, i dati relativi a S146 e D146 sono stati ottenuti principalmente a Cipro. Dal momento che non ci sono dati riguardo a questi due alleli in altri paesi europei a causa della loro assenza o bassa frequenza, non è possibile asserire che questi polimorfismi siano responsabili della resistenza nei confronti di altri ceppi di scrapie circolanti in Europa. Nella popolazione caprina italiana sono stati trovati i seguenti polimorfismi: G37V, P42P, T110P, G127S, L133Q, M137I, S138S, I142M, I142T, H143R, R151H, R154H, P168Q, T194P, T202T, R211Q, T219I, Q222K, G232G, S240P (Colussi et al., 2010, Curcio, 2016, Vaccari et al., 2009); inoltre, sono state trovate differenze importanti tra la frequenza dell'allele K222 in razze caprine allevate nel nord Italia (1-5%) con razze caprine allevate nel sud Italia (15-18%) (Acutis et al., 2008). Lo scopo di questo lavoro è stato quello di esaminare i polimorfismi a carico del gene PRNP in 10 razze caprine allevate nell'Italia centro meridionale (Camosciata delle Alpi, Saanen, Grigia Ciociara, Bianca Monticellana, Capestrina, Fulva del Lazio, Facciuta della Valnerina, Teramana, Garganica, Grigia Molisana e meticci) al fine di valutarne la distribuzione sul territorio.
Materiali e metodi
Sono stati prelevati campioni di sangue provenienti da quattro razze/popolazioni allevate nel Lazio e in Campania: Grigia Ciociara (24), Bianca Monticellana (24), Capestrina (23), Fulva del Lazio (24), Facciuta della Valnerina (24) allevata in Umbria, Teramana (24) allevata in Abruzzo, Garganica (24) allevata in Puglia, Basilicata, Campania e Calabria, Grigia Molisana (24) allevata in Molise e due razze cosmopolite Camosciata delle alpi (203) e Saanen (20). Inoltre, è stato anche campionato un gruppo di capre meticce (51) provenienti dalle stesse zone. Dal sangue è stato estratto il DNA genomico ed è stato amplificato per PCR il gene PRNP (Bossers et al., 1996), dopo purificazione, l'amplificato è stato sequenziato bidirezionalmente e le sequenze sono state sottoposte ad analisi bioinformatica per l'allineamento con software BioEdit v 7.2.5.
Risultati e discussione
I polimorfismi rilevati, che portano ad una sostituzione amminoacidica, sono stati osservati in 15 codoni: G37V, T110P, G127S, M137I, I142M/I142 T, H143R, N146S, R154H, P168Q, T194P, R211Q, Q215R, Q220H, Q222K, S240P. Sono state anche rilevate 2 mutazioni silenti ai codoni 42 (ccg/cca) su 220 capre e 138 (agc/agt) su 210 capre. (Tabella 1)
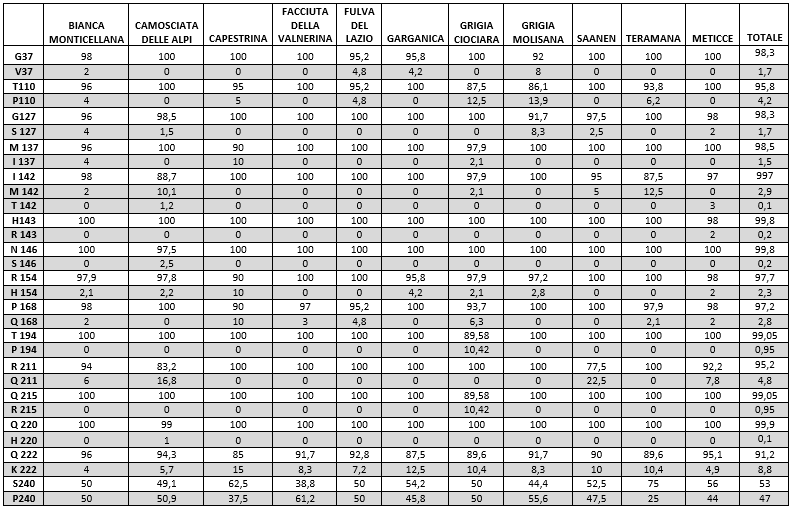
Table 1. Percentage of allele frequencies divided by race (in the gray rows mutated codons)
Mediamente varianti alleliche sono poco rappresentate, da 0,1 % (T142-H 220) al 4,8 % (Q 211), eccetto per l'allele K222 (8,8%) e P240 (47%). La percentuale dell'allele K222 è risultata del 4% per la Bianca Monticellana, 5,7% per la Camosciata delle alpi con due soggetti in omozigosi, 15% per la Capestrina, 8,3% per la Facciuta della Valnerina con un soggetto in omozigosi, 7,2% per la Fulva del Lazio con un soggetto in omozigosi, 12,5% per la Garganica con un soggetto in omozigosi, 10,4% per la Grigia Ciociara, 8,3% per la Grigia Molisana, 10% per la Saanen, 10,4% per la Teramana, e 4,9% per un gruppo di meticce. Riguardo le altre varianti che rendono gli animali meno suscettibili: S127, M142, T142 R143, H154 e Q211, la frequenza allelica globale è risultata pari a:1,7%, 2,9%, 0,1%, 0,2%, 2,3% e 4,8 %.
Sorprendentemente è stata riscontrata una frequenza allelica del 146S pari al 2,5% nella razza Camosciata delle Alpi. Tale osservazione rappresenta la prima descrizione di questa variante in Italia.
Conclusioni
I risultati ottenuti potrebbero essere utili per la progettazione di un piano di selezione genetica.
Infatti, per tutte le razze analizzate sono presenti con varie frequenze animali portatori dell'allele K222 che potrebbero essere utilizzati in un programma di selezione genetica volto ad aumentare la resistenza alla scrapie classica nella popolazione caprina italiana.
Bibliografia
Acutis PL, Bossers A, Priem J, Riina MV, Peletto S, et al. (2006) Identification of prion protein gene polymorphisms in goats from Italian scrapie outbreaks. J Gen Virol 87: 1029-1033.
Acutis 2008 Acutis P.L., Colussi S., Santagata G., Laurenza M.C. Maniaci M.G., Riina M.V., Peletto S., Goldmann W., Bossers A.,Caramelli M., Cristoferi I.,Maimone S.,Sacchi P., e Rasero R.. (2008) Genetic variability of PRNP gene in goat breed from Northern and Southern Italy. J. Appl. Microbiol. 104: 1782-1789
Agrimi U., Conte M., Morelli L., Di Bari MA., Di Guardo G., Ligios C., Antonucci G., Aufiero G., Pozzato N., Mutinelli F., Nonno R. e Vaccari G.. (2003) Animal transmissible spongiform encephalopathies and genetics. Veterinary Research Communications 27: 31-38.
Aguilar-Calvo P., Espinosa J.C., Pintado B., Gutiérrez-Adán A., Alamillo E., Miranda A., Prieto I., Bossers A., Andreoletti O., e Torres J.M. (2014) Role of the goat K222-PrPC polymorphic variant in prion infection resistance. Journal of Virology 88: 2670-2676.
Aguzzi A., Heikenwalder M. e Polymenidou M. (2007). Insights into prion strains and neurotoxicity. Nature Reviews Molecular Cell Biology 8: 552-561.
Babelhadj B, Di Bari MA, Pirisinu L, Chiappini B, Gaouar SBS, Riccardi G, Marcon S, Agrimi U, Nonno R, Vaccari G. (2018) Prion Disease in Dromedary Camels, Algeria. Emerg Infect Dis. 24(6):1029-1036.
Barillet F., Mariat D., Amigues Y., Faugeras R., Caillat H., et al. (2009) Identification of seven haplotypes of the caprine PrP gene at codons 127, 142, 154, 211, 222 and 240 in French Alpine and Saanen breeds and their association with classical scrapie. J Gen Virol 90: 769-776.
Baylis M. e Goldmann W. (2004). The genetics of scrapie in sheep and goats. Current Molecular Medicine 4: 385-396.
Billinis C., Panagiotidis C.H., Psychas V., Argyroudis S., Nicolaou A., et al. (2002) Prion protein gene polymorphisms in natural goat scrapie. J Gen Virol 83: 713-721.
Bossers A., Screuder B.E.C., Muileman I.H., Belt P.B., e Smits M.A. (1996) PrP genotypecontributes to determining survivaltimes of sheepwith natural scrapie. J. Gen. Virol. 77: 2669-2673
Cassard H., Torres J.M., Lacroux C., Douet J.Y., Benestad S.L., Lantier F., Lugan F., Lantier I., Costes P., Aron N., Reine F., Herzog L., Espinosa J.C., Beringue V. e Andréoletti O. (2014) Evidence for zoonotic potential of ovine scrapie prions. Nature Communications 5: 5821-5829.
Colussi S., Vaccari G., Rasero R., Ponti A.M., Ru G., Sacchi P., Caramelli M., Agrimi U. e Acutis PL. (2010). Prospects for applying breeding for resistance to control scrapie in goats: The current situation in Italy. Small Ruminant Research 88: 97-101.
Colussi S., Vaccari G., Maurella C., Bona C., Lorenzetti R., Troiano P., Casalinuovo F., Di Sarno A., Maniaci MG., Zuccon F., Nonno R., Casalone C., Mazza M., Ru G.,Caramelli M., Agrimi U., Acutis PL.(2008) Histidine at codon 154 of the prion protein gene is a risk factor for NOR 98 scrapie in goats. J:Gen. Virol. 89: 3173-3176
Commission Decision 2003/100/EC 2003. Commission Decision of laying down minimum requirements for the establishment of breeding programmes for resistance to transmissible spongiform encephalopathies in sheep. Official Journal of the European Union L41, 41-43. Retrieved on 27 September 2015 from http://eur-lex.europa.eu/legal-content/EN/TXT/?uri=uriserv: OJ.L_.2003.041.01.0041.01.ENG.
Comoy EE., Mikol J., Luccantoni-Freire S., Correia E., Lescoutra-Etchegaray N., Durand V., Dehen C., Andréoletti O., Casalone C., Richt JA., Greenlee JJ., Baron T., Benestad SL., Brown P. e Deslys JP. (2015) Transmission of scrapie prions to primate after an extended silent incubation period. Scientific Reports 5: 11573-11583.
Curcio L., Sebastiani C., Di Lorenzo P., Lasagna E e Biagetti M. (2016) Review: A review on classical and atypical scrapie in caprine: Prion protein gene polymorphisms and their role in the disease. Animal. 10:1585-1593
European Food Safety Authority (EFSA) (2017) Genetic resistance to transmissible spongiform encephalopathies (TSE) in goats. EFSA Journal 15(8):4962
Fast C., Golmann W., Berthon P., Tauscher K., Andreoletti O., Lantier I., Rossignol C., Bossers A., Jacobs JG., Hunter N., Groschup MH., Lantier F., Langeveld JPM. (2017) Protecting effect of PrP codons M142 and K 222 in goats orally challenged with bovine spongiform encephalopathy prions. Vet.res. 48: 55.
Fragkiadaki EG., Vaccari G., Ekateriniadou LV., Agrimi U., Giadinis ND., Chiappini B., Esposito E., Conte M., Nonno R. (2011) PRNP genetic variability and molecular typing of natural goat scrapie isolates in a high number of infected flocks. Vet Res 42:104.
Glatzel M. e Aguzzi A. (2001) The shifting biology of prions. Brain Research Review 36: 241-248
Goldman W. (2008) PrP Genetics in ruminant transmissible spongiform encephalopathies. Veterinary research 39: 1-14
Goldmann W., Martin T., Foster J., Hughes S., Smith G., et al. (1996) Novel polymorphisms in the caprine PrP gene: a codon 142 mutation associated with scrapie incubation period. J. Gen. Virol. 77: 2885-2891
Goldmann W., Ryan K., Stewart P., Parnham D., Xicohtencatl R., et al. (2011) Caprine prion gene polymorphisms are associated with decreased incidence of classical scrapie in goat herds in the United Kingdom. Vet Res 42: 110-117.
Hunter N. (1997) PrP genetics in sheep and the implications for scrapie and BSE. Trends in Microbiology 5: 331-334
Papasavva-Stylianou P., Kleanthous M., Toumazos P., Mavrikiou P., Loucaides P. (2007) Novel polymorphisms at codons 146 and 151 in the prion protein gene of Cyprus goats, and their association with natural scrapie. Vet. J. 173:459-462.
Papasavva-Stylianou P., Windl O., Saunders G., Mavrikiou P., Toumazos P., et al. (2011) PrP gene polymorphisms in Cyprus goats and their association with resistance or susceptibility to natural scrapie. Vet J 187: 245-250.
Prusiner SB. (2013) Biology and genetics of prions causing neurodegeneration. Annual Review of Genetics 47: 601-623
Prusiner SB. (1991) Molecular biology of prion diseases. Science 252: 1515-1522.
Srithayakumar V., Mitchell GB., White BN. (2016) Identification of amino acid variation in the prion protein associated with classical scrapie in Canadian dairy goats BMC Vet Res. 22:12-59.
Vaccari G., Di Bari MA., Morelli L., Nonno R., Chiappini B., et al. (2006) Identification of an allelic variant of the goat PrP gene associated with resistance to scrapie. J Gen Virol 87: 1395-1402.
Vaccari G., Panagiotidis C., Acín C., Peletto S., Barillet F., Acutis PL., Bossers A., Langeveld J., van Keulen L., Sklaviadis T., Badiola JJ., Andreoletti O., Groschup MH., Agrimi U., Foster J., Goldmann W. (2009) State of the art review of goat TSE in the European Union with special emphasis on PRNP genetics and epidemiology. Vet Res 40:48.
Acutis 2008 Acutis P.L., Colussi S., Santagata G., Laurenza M.C. Maniaci M.G., Riina M.V., Peletto S., Goldmann W., Bossers A.,Caramelli M., Cristoferi I.,Maimone S.,Sacchi P., e Rasero R.. (2008) Genetic variability of PRNP gene in goat breed from Northern and Southern Italy. J. Appl. Microbiol. 104: 1782-1789
Agrimi U., Conte M., Morelli L., Di Bari MA., Di Guardo G., Ligios C., Antonucci G., Aufiero G., Pozzato N., Mutinelli F., Nonno R. e Vaccari G.. (2003) Animal transmissible spongiform encephalopathies and genetics. Veterinary Research Communications 27: 31-38.
Aguilar-Calvo P., Espinosa J.C., Pintado B., Gutiérrez-Adán A., Alamillo E., Miranda A., Prieto I., Bossers A., Andreoletti O., e Torres J.M. (2014) Role of the goat K222-PrPC polymorphic variant in prion infection resistance. Journal of Virology 88: 2670-2676.
Aguzzi A., Heikenwalder M. e Polymenidou M. (2007). Insights into prion strains and neurotoxicity. Nature Reviews Molecular Cell Biology 8: 552-561.
Babelhadj B, Di Bari MA, Pirisinu L, Chiappini B, Gaouar SBS, Riccardi G, Marcon S, Agrimi U, Nonno R, Vaccari G. (2018) Prion Disease in Dromedary Camels, Algeria. Emerg Infect Dis. 24(6):1029-1036.
Barillet F., Mariat D., Amigues Y., Faugeras R., Caillat H., et al. (2009) Identification of seven haplotypes of the caprine PrP gene at codons 127, 142, 154, 211, 222 and 240 in French Alpine and Saanen breeds and their association with classical scrapie. J Gen Virol 90: 769-776.
Baylis M. e Goldmann W. (2004). The genetics of scrapie in sheep and goats. Current Molecular Medicine 4: 385-396.
Billinis C., Panagiotidis C.H., Psychas V., Argyroudis S., Nicolaou A., et al. (2002) Prion protein gene polymorphisms in natural goat scrapie. J Gen Virol 83: 713-721.
Bossers A., Screuder B.E.C., Muileman I.H., Belt P.B., e Smits M.A. (1996) PrP genotypecontributes to determining survivaltimes of sheepwith natural scrapie. J. Gen. Virol. 77: 2669-2673
Cassard H., Torres J.M., Lacroux C., Douet J.Y., Benestad S.L., Lantier F., Lugan F., Lantier I., Costes P., Aron N., Reine F., Herzog L., Espinosa J.C., Beringue V. e Andréoletti O. (2014) Evidence for zoonotic potential of ovine scrapie prions. Nature Communications 5: 5821-5829.
Colussi S., Vaccari G., Rasero R., Ponti A.M., Ru G., Sacchi P., Caramelli M., Agrimi U. e Acutis PL. (2010). Prospects for applying breeding for resistance to control scrapie in goats: The current situation in Italy. Small Ruminant Research 88: 97-101.
Colussi S., Vaccari G., Maurella C., Bona C., Lorenzetti R., Troiano P., Casalinuovo F., Di Sarno A., Maniaci MG., Zuccon F., Nonno R., Casalone C., Mazza M., Ru G.,Caramelli M., Agrimi U., Acutis PL.(2008) Histidine at codon 154 of the prion protein gene is a risk factor for NOR 98 scrapie in goats. J:Gen. Virol. 89: 3173-3176
Commission Decision 2003/100/EC 2003. Commission Decision of laying down minimum requirements for the establishment of breeding programmes for resistance to transmissible spongiform encephalopathies in sheep. Official Journal of the European Union L41, 41-43. Retrieved on 27 September 2015 from http://eur-lex.europa.eu/legal-content/EN/TXT/?uri=uriserv: OJ.L_.2003.041.01.0041.01.ENG.
Comoy EE., Mikol J., Luccantoni-Freire S., Correia E., Lescoutra-Etchegaray N., Durand V., Dehen C., Andréoletti O., Casalone C., Richt JA., Greenlee JJ., Baron T., Benestad SL., Brown P. e Deslys JP. (2015) Transmission of scrapie prions to primate after an extended silent incubation period. Scientific Reports 5: 11573-11583.
Curcio L., Sebastiani C., Di Lorenzo P., Lasagna E e Biagetti M. (2016) Review: A review on classical and atypical scrapie in caprine: Prion protein gene polymorphisms and their role in the disease. Animal. 10:1585-1593
European Food Safety Authority (EFSA) (2017) Genetic resistance to transmissible spongiform encephalopathies (TSE) in goats. EFSA Journal 15(8):4962
Fast C., Golmann W., Berthon P., Tauscher K., Andreoletti O., Lantier I., Rossignol C., Bossers A., Jacobs JG., Hunter N., Groschup MH., Lantier F., Langeveld JPM. (2017) Protecting effect of PrP codons M142 and K 222 in goats orally challenged with bovine spongiform encephalopathy prions. Vet.res. 48: 55.
Fragkiadaki EG., Vaccari G., Ekateriniadou LV., Agrimi U., Giadinis ND., Chiappini B., Esposito E., Conte M., Nonno R. (2011) PRNP genetic variability and molecular typing of natural goat scrapie isolates in a high number of infected flocks. Vet Res 42:104.
Glatzel M. e Aguzzi A. (2001) The shifting biology of prions. Brain Research Review 36: 241-248
Goldman W. (2008) PrP Genetics in ruminant transmissible spongiform encephalopathies. Veterinary research 39: 1-14
Goldmann W., Martin T., Foster J., Hughes S., Smith G., et al. (1996) Novel polymorphisms in the caprine PrP gene: a codon 142 mutation associated with scrapie incubation period. J. Gen. Virol. 77: 2885-2891
Goldmann W., Ryan K., Stewart P., Parnham D., Xicohtencatl R., et al. (2011) Caprine prion gene polymorphisms are associated with decreased incidence of classical scrapie in goat herds in the United Kingdom. Vet Res 42: 110-117.
Hunter N. (1997) PrP genetics in sheep and the implications for scrapie and BSE. Trends in Microbiology 5: 331-334
Papasavva-Stylianou P., Kleanthous M., Toumazos P., Mavrikiou P., Loucaides P. (2007) Novel polymorphisms at codons 146 and 151 in the prion protein gene of Cyprus goats, and their association with natural scrapie. Vet. J. 173:459-462.
Papasavva-Stylianou P., Windl O., Saunders G., Mavrikiou P., Toumazos P., et al. (2011) PrP gene polymorphisms in Cyprus goats and their association with resistance or susceptibility to natural scrapie. Vet J 187: 245-250.
Prusiner SB. (2013) Biology and genetics of prions causing neurodegeneration. Annual Review of Genetics 47: 601-623
Prusiner SB. (1991) Molecular biology of prion diseases. Science 252: 1515-1522.
Srithayakumar V., Mitchell GB., White BN. (2016) Identification of amino acid variation in the prion protein associated with classical scrapie in Canadian dairy goats BMC Vet Res. 22:12-59.
Vaccari G., Di Bari MA., Morelli L., Nonno R., Chiappini B., et al. (2006) Identification of an allelic variant of the goat PrP gene associated with resistance to scrapie. J Gen Virol 87: 1395-1402.
Vaccari G., Panagiotidis C., Acín C., Peletto S., Barillet F., Acutis PL., Bossers A., Langeveld J., van Keulen L., Sklaviadis T., Badiola JJ., Andreoletti O., Groschup MH., Agrimi U., Foster J., Goldmann W. (2009) State of the art review of goat TSE in the European Union with special emphasis on PRNP genetics and epidemiology. Vet Res 40:48.
RICERCA FINANZIATA DAL MINISTERO DELLA SALUTE RC004/2015

Torricelli 2019 - Fattori genetici di resistenza/suscettibilitą alla Scrapie Classica nella specie caprina (SPVet.it 113/2019)

Fattori genetici di resistenza/suscettibilitą alla Scrapie Classica nella specie caprina by Torricelli et al., 2019 is licensed under a Creative Commons Attribution 4.0 International License.
Permissions beyond the scope of this license may be available at http://indice.spvet.it/adv.html.