Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Webzine Sanità Pubblica Veterinaria: Numero 31, luglio - ottobre 2005 [http://spvet.it]Documento reperibile all'indirizzo: http://spvet.it/indice.html#290
The real time PCR for bovine leukemia virus diagnosis.
Impiego della real time PCR per la diagnosi della Leucosi bovina
enzootica.
De Giuseppe A., Feliziani F., Giammarioli M., Marchesi M.C., Marini C., Rutili D. e De Mia G.M.
Abstract: Sensitive assays are required to detect proviral bovine leukemia virus (BLV) in blood or tissue samples from cattle. A Real-Time (TaqMan) PCR, conducted using primers and
TaqMan probe designed to amplify and specifically detect a 122 bp fragment of BLV gp30 envelope was developed and compared to the conventional enzyme immunosorbent assay
(ELISA). For this study, field samples (n=141) from cattle coming from several Italian regions were employed. The Real-time (TaqMan) PCR identified 98% of buffy coats from BLV
seropositive cattle (n=53) whereas no amplification was observed in all the seronegative samples (n=88). These results showed a good correlation between Real-time (TaqMan)
PCR and ELISA (=99,3%).
INTRODUZIONE
Tra le patologie infettive dei ruminanti sostenute da Retrovirus, la Leucosi Enzootica Bovina (LEB) gioca senza dubbio un ruolo d'importanza rilevante. La LEB è una malattia
contagiosa dei bovini sostenuta da un virus la cui replicazione non viene bloccata dalla risposta anticorpale umorale dell'ospite. La eradicazione della LEB è stata resa obbligatoria su
tutto il territorio nazionale a partire dal 1996 ed i progressi ottenuti nel controllo della malattia sono testimoniati dalle basse prevalenze d'infezione riscontrate in questi ultimi anni.
Restano tuttavia nel territorio "code" di eradicazione la cui eliminazione appare più problematica del previsto, contribuendo a porre il livello sanitario del nostro paese su un piano
decisamente inferiore rispetto a quello di altri paesi dell'Unione europea.
Le tradizionali metodiche diagnostiche comunemente impiegate per le infezioni sostenute da Retrovirus, si basano essenzialmente sulla sierologia. Per quanto riguarda la LEB, nel
corso degli ultimi anni sono andate sviluppandosi anche metodiche diagnostiche biomolecolari (1, 2, 4, 5) che trovano, tra l'altro, un'utile applicazione nella diagnosi della forma
tumorale, attualmente eseguita con l'esame istologico, che non consente però di differenziare i diversi tipi di linfosarcoma riscontrabili nelle forme di leucosi enzootica e sporadica.
Alla luce di quanto sopra esposto, con il presente lavoro, si illustrano i risultati di un'attività di ricerca volta a mettere a punto e validare l'evidenziazione dell'acido nucleico del virus
della LEB attraverso la Real-Time PCR; si ritiene infatti che questa tecnica possa costituire un valido ausilio diagnostico, in particolare, per la sua sensibilità analitica.
MATERIALI E METODI
Campioni esaminati
Colture cellulari della linea continua Foetal Lamb Kidney (FLK) vergini o permanentemente infette con il virus LEB, sono state utilizzate rispettivamente come
campioni negativo e positivo nella standardizzazione della Real-Time PCR.
Per la verifica della sensibilità analitica della metodica, oltre alle FLK infette sono stati impiegati campioni di sangue (buffy coats) provenienti da tre bovini sperimentalmente infettati.
Il DNA genomico estratto è stato quantificato mediante spettrofotometro e sottoposto a diluizioni logaritmiche. Ogni diluizione è stata processata in tre repliche.
Sono anche stati saggiati 53 buffy coats provenienti da campioni di sangue raccolti da bovini risultati positivi al test ELISA. I bovini postivi sono stati individuati nell'ambito del piano
nazionale di eradicazione nei confronti della LEB in diversi allevamenti sparsi nel territorio italiano. Sono stati inoltre esaminati 88 buffy coats provenienti da campioni di sangue
raccolti da bovini risultati negativi ai tests sierologici e provenienti da allevamenti ufficialmente indenni da LEB.
Scelta dei primers e della sonda TaqMan. Per la messa a punto del test Real-time PCR, è stata individuata la sequenza nucleotidica da amplificare in una regione altamente
conservata del genoma virale codificante la glicoproteina gp30 dell' envelope del virus della LEB (3).
Le sequenze degli oligonucleotidi disegnati per l'amplificazione di un target di 122 bp della gp30, sono le seguenti:
5'-TTAGGCCTAGCCCTGTCAGTG-3' (forward primer)
5'-TGCTGTGATCAAGCGTTGCTG-3' (reverse primer).
La seguente sonda TaqMan, è stata disegnata per rilevare le sequenze target specifiche nei campioni di cDNA amplificato:
5'-FAM-ATCAGAGACTCACCTCCCTGATCCACGTTC-
-T-TAMRA-3'.
Estrazione del DNA genomico
I campioni in esame sono stati sottoposti ad estrazione del DNA genomico mediante l'impiego del Kit QIAamp DNA Mini Kit (Qiagen) a partire da 30 l
di materiale (buffy coats o FLK) diluiti in 170 l tampone PBS, secondo la procedura riportata nel protocollo del Kit.
Real-Time PCR (TaqMan)
La prova è stata eseguita con la macchina ABI PRISM 7700 (Applied Biosystem), nelle seguenti condizioni: PCR Gold buffer 1x (Roche-Perkin Elmer),
MgCl 2.5 mM, dNTP 200 M, 20 pmoli di primers, 12.5 pmoli di sonda TaqMan, 2.5 U di AmpliTaq Gold e 100 ng di DNA in un volume totale di 50 l. L'amplificazione ha previsto 1 ciclo a 2
95 °C x 10', denaturazione a 95 °C x 15'', annealing a 65 °C x 1'ed estensione a 72 °C x 1' per un totale di 40 cicli.
RISULTATI
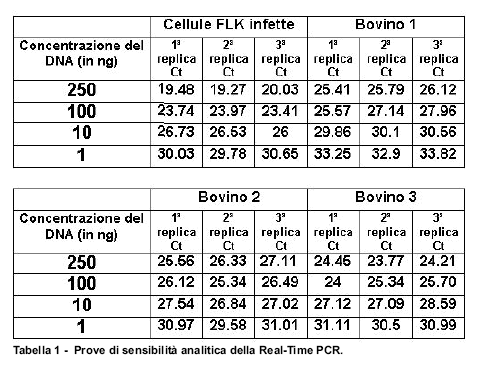
Nella tabella 1 sono riportati i valori del ciclo soglia (Ct) ottenuti dalle prove effettuate
per la valutazione della sensibilità della Real-Time PCR. Per un più facile confronto
dei risultati ottenuti, nel grafico 1 si riportano i valori medi dei Ct delle tre repliche per
ogni campione esaminato.
Tutte e tre le repliche hanno dato esito positivo sino alla
diluizione massima testata (1 ng di DNA genomico). Sulla base di questi risultati ed
anche in considerazione di una prassi oramai consolidata circa la concentrazione di
DNA da impiegare nella Real-Time PCR, tutte le prove di amplificazione sono state
eseguite utilizzando 100 ng di DNA estratto dai campioni in esame.
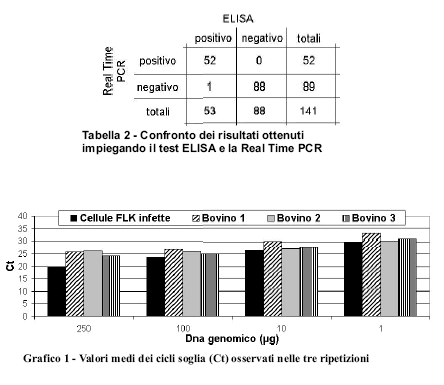
Dai risultati ottenuti è possibile stimare i valori di concordanza tra i due metodi. La
proporzione di concordanza osservata è pari al 99,3%. Applicando il test Kappa ai dati
in nostro possesso, si ottiene un valore molto alto (K=0,985) con una differenza non
statisticamente significativa dalla concordanza assoluta (IC 0,955-1,014 con LC
95%).
CONCLUSIONI
Scopo del presente lavoro è stato quello di mettere a punto e validare una metodica diagnostica biomolecolare per la diagnosi di LEB, la Real Time (TaqMan) PCR, con il
presupposto che avrebbe presentato, rispetto ai metodi diagnostici tradizionali, numerosi vantaggi. In primo luogo quello di offrire la possibilità di evidenziare gli stadi precoci
dell'infezione e, conseguentemente, risultare vantaggiosa proprio nella eliminazione di quelle "code" di eradicazione cui si è accennato in introduzione. La prova è stata messa
a punto disegnando una coppia di primers diretta verso una regione altamente conservata del genoma virale. In mancanza di un gold standard la prova di riferimento non poteva
che essere di tipo sierologico (ELISA).
I valori di concordanza osservati tra le due prove si sono rivelati molto alti e inducono a ritenere che la Real Time PCR sia caratterizzata
da performances non dissimili da quelle dell'ELISA. Per una verifica più approfondita dei parametri di sensibilità e specificità sarebbe però necessario un campionamento
diverso, soprattutto per quel che riguarda la numerosità.
Infatti, riguardo alla sensibilità analitica, ci si attende un salto di qualità, anche in considerazione della elevata
sensibilità “intrinseca” mostrata dalla Real Time PCR per la diagnosi di altre patologie infettive Va anche detto che, una cosa è mettere in evidenza il virus (e il suo genoma),
altra cosa ricercare, come nel caso della LEB, il provirus il cui genoma è presente negli organi e tessuti target in forma integrata. Da questo punto di vista, i nostri risultati non si
discostano sostanzialmente da quanto riportato in letteratura (2, 3). D'altra parte, l'impiego della sonda TaqMan, specifica per LEB, garantisce alla prova caratteristiche tali di
specificità che nessun sistema immunoenzimatico può, al momento, vantare.
In aggiunta, può trovare un utile impiego, come detto, nella diagnosi delle forme tumorali e può
rappresentare un ottimo ausilio nella conferma di casi che abbiano fornito risultati sierologici non conclusivi. La Real Time PCR potrebbe infatti essere utilmente impiegata nei
casi di sieropositività sporadiche che, talvolta, si riscontrano in allevamenti da tempo ufficialmente indenni. Per quanto riguarda invece il suo eventuale impiego routinario nel
corso di campagne profilattiche, riteniamo che, al momento, non rappresenti un'alternativa valida ai presidi attualmente in uso, in special modo per i costi che comporterebbe.
BIBLIOGRAFIA
1. Gutierrez S.E., Dolcini D.L., Arroyo G.H., Rodriguez-Dubra C., Ferrer J.F., Esteban E.N., 2001. Development and evaluation of highly sensitive and specific blocking enzyme-linked immunosorbent assay and polymerase chain reaction assay for diagnosis of bovine leukemia virus infection
in cattle. American Journal of Veterinary Research 62: 10, 1571-1577.
2. Kuckleburg C.J., Chase C.C., Nelson E.A., Marras S.A.E., Dammen M.A., Christopher-Hennings J., 2003. Detection of bovine leukemia virus in blood and milk by nested and real-time polymerase chain reactions. Journal of Veterinary Diagnostic Investigation 15: 1, 72-76.
3. Mamoun R.Z., Morrison M., Rebeyrotte N., Busetta B., Couez D., Ketteman R., Hospital M., and Guilleman B., 1990. Sequence Variability of Bovine Leukemia Virus env Gene and Its Relevance to the Structure and Antigenecity of the Glycoproteins. Journal of Virology, 64: 9, 4180-4188.
4. Markiewicz L., Rulka J., Kaminski S., 2003. Detection of BLV provirus in different cells by nested-PCR. Bulletin of the Veterinary Institute in Pulawy, 47: 2, 325-331.
5. Nagy D.W., Tyler J.W., Kleiboeker S.B., Stoker A., 2003. Use of polymerase chain reaction assay to detect bovine leukosis virus in dairy cattle. Journal of the American Veterinary Medical Association 222: 7, 983-985.
---------------------------
Lavoro presentato al SIDILV - SOCIETA' ITALIANA DI DIAGNOSTICA DI LABORATORIO VETERINARIA VI Congresso Nazionale, Abano Terme (PD), 10-12 novembre 2004
Ricerca finanziata dal Ministero della Salute, (DL 502/92, art. 12), RC 2002 IZSUM 05/2002