Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Webzine Sanità Pubblica Veterinaria: Numero 63, Dicembre 2010 [http://www.spvet.it/] ISSN 1592-1581
Caratterizzazione molecolare e distribuzione geografica di M. bovis e M. caprae isolati in Umbria e Marche tra il 2002 e il 2007 - Geographical distribution and molecular characterization of M. bovis and M. caprae isolated in Umbria and Marche Regions (Italy) between 2002 and 2007
Mazzone P., Boniotti M.B., Biagetti M., Crotti S., Ciullo M., Cagiola M., Corneli S., Duranti A., Faccenda L., Gavaudan S., Pacciarini M. L.
Abstract. Mycobacterium bovis and Mycobacterium caprae are the causative agents of bovine tuberculosis (TB), disease that still representing a serious concern both for its health implications and economic impact. The use of an integrated approach, combining epidemiology and molecular biology, may be a support in the eradication plans, allowing to trace the origins and modes of transmission of TB outbreaks and to implement control measures to adapt to different local situations. Since 2002 the Institute Zooprofilattico Umbria and Marche, in collaboration with the National Reference Centre, has carried out genotyping of strains of M. bovis and M. caprae isolated from outbreaks of TB, in order to define the epidemiology of TB in the two regions and to provide useful elements for the achievement of the official TB-free status. Furthermore, following the detection of M. bovis in wild boar, a survey was conducted to assess the prevalence of M. bovis in wild populations in the two regions. These activities allowed us to genotype strains of M. bovis isolates in wild boars and to perform a comparison with the genetic profiles of isolates in cattle. The molecular typing techniques used were Spoligotyping and analysis of ETRs loci (Exact Tandem Repeats). The 61 strains of M. bovis and M. caprae (50 strains isolated from cattle outbreaks in the two regions and 11 strains isolated from wild boars in Marche) were discriminated in 8 spoligotypes, including M. bovis SB0120, common in Umbria and Marche and M. caprae, present only in Umbria. The 8 spoligotypes were further differentiated by ETRs analysis into 21 genotypes (11 located in Umbria, 7 in Marche and 3 localized in both regions). Among the strains circulating in the Marche region, the "M. bovis SB0120-ETRs 33533" isolated from cattle was also found in nine wild boars from the same territory. About M. caprae, the genotype SB0418, combined with ETRs 63525, 53535 and 53525, was found in Umbria and in outbreaks that occurred in the same period in farms of South Italy. The tracing of animal movements allowed us to identify a single geographic area as possible origin of the infected animals. Moreover the genetic characterization of M. caprae isolates from outbreaks in Umbria has allowed us to trace back to the same territory, supporting the hypothesis of conventional epidemiological investigations. Spoligotyping has been described as a technique poorly discriminating and it was crucial the integration with ETR analysis to reveal interesting homologies between the circulating strains. The genotyping of isolates and the geographical location of outbreaks allowed us also to detect the presence of M. bovis strains with the same genetic profiles isolated in outbreaks occurring between the two regions; this observation led us to extend epidemiological investigations to clarify the transmission routes of the disease in the examined territory.
Riassunto. Mycobacterium bovis e Mycobacterium caprae sono gli agenti causali della tubercolosi bovina (TB), malattia che costituisce ancora un serio problema sia per le implicazioni di carattere sanitario che per le importanti ripercussioni economiche. L'adozione di un approccio integrato, che combini l'epidemiologia con la biologia molecolare, permette di supportare i Piani di eradicazione, di comprendere le origini e le modalità di trasmissione della TB e di attivare misure di controllo da adattare alle diverse realtà territoriali. L'Istituto Zooprofilattico dell'Umbria e delle Marche, per meglio definire l'epidemiologia della TB nelle due regioni e nell'ottica di fornire elementi utili per il raggiungimento della qualifica europea nel territorio di competenza, dal 2002 ha effettuato, in collaborazione con il Centro Nazionale di Referenza per la Tubercolosi bovina, la genotipizzazione dei ceppi di M. bovis e M. caprae isolati nei focolai di TB. Contestualmente, in seguito al riscontro di alcune positività per M. bovis nei cinghiali, è stata condotta un'indagine conoscitiva per valutare la diffusione del M. bovis nelle popolazioni selvatiche presenti nelle due regioni; tale attività ha permesso di genotipizzare anche i ceppi di M. bovis isolati nei cinghiali e di eseguire un confronto con i profili genetici degli isolati nei bovini. Le tecniche di tipizzazione molecolare adottate sono state lo Spoligotyping e l'analisi dei loci ETRs (Exact Tandem Repeats). I 61 ceppi di M. bovis e M. caprae tipizzati (50 isolati da focolai bovini delle due regioni e 11 isolati nei cinghiali delle Marche) sono stati distinti in 8 spoligotipi, comprendenti M. bovis SB0120, diffuso in Umbria e Marche e M. caprae, presente solo in Umbria. Gli 8 spoligotipi sono stati ulteriormente differenziati con il saggio ETRs in 21 sottogruppi (11 localizzati in Umbria, 7 nelle Marche e 3 presenti in entrambe le regioni). Dei ceppi circolanti nel territorio marchigiano, il Mycobacterium bovis SB0120 ETRs 33533 isolato dai bovini è stato rinvenuto anche in 9 cinghiali provenienti dalla stessa località. Nel caso dei ceppi di M. caprae, il genotipo SB0418, combinato con gli ETRs 63525, 53535 e 53525, oltre che in Umbria, è stato trovato quasi esclusivamente in focolai che si sono verificati, nello stesso arco di tempo, in aziende del Sud del nostro paese. La ricostruzione della movimentazione degli animali ha permesso d'individuare un'unica area geografica come possibile zona di origine dei capi infetti, allo stesso tempo, la caratterizzazione genetica dei ceppi di M. caprae isolati nei focolai umbri ha consentito di risalire al medesimo territorio, avvalorando le ipotesi delle indagini convenzionali. Come descritto in letteratura lo Spoligotyping si è rivelato una tecnica scarsamente discriminante ed è stato determinante affiancare l'analisi dei loci ETRs per svelare interessanti omologie tra i ceppi circolanti. La genotipizzazione degli isolati e la localizzazione geografica dei focolai hanno permesso anche di evidenziare la presenza di M. bovis con profili genetici uguali, isolati in focolai a cavallo tra le due regioni; tale osservazione induce ad approfondire ulteriormente l'indagine epidemiologica per chiarire le modalità di diffusione della malattia nel territorio considerato.
Introduzione
I programmi di eradicazione della tubercolosi bovina applicati nei paesi industrializzati si basano principalmente sul test intradermico singolo e comparativo (IDT), sulla macellazione degli animali infetti e il successivo isolamento del micobatterio coinvolto. In un secondo tempo, assolti i doveri normativi, può essere utile eseguire la genotipizzazione dei ceppi di Mycobacterium bovis e Mycobacterium caprae isolati nell'ambito dei diversi focolai, al fine di combinare le informazioni ottenute dalla biologia molecolare con le informazioni dell'indagine epidemiologica tradizionale.
Questo tipo di approccio integrato permette di supportare i piani di eradicazione, di comprendere le origini e le modalità di trasmissione della TB e di attivare misure di controllo da adattare alle diverse realtà territoriali.
L'Istituto Zooprofilattico dell'Umbria e delle Marche, per meglio definire l'epidemiologia della malattia nel territorio di competenza e nell'ottica di fornire elementi utili per il raggiungimento della qualifica europea (DM 592 del 15 Dicembre 1995, D.Lgs. 196 del 22 maggio 1999), dal 2002 ha effettuato, in collaborazione con il Centro di Referenza Nazionale per la Tubercolosi bovina da M. bovis, presso l'IZS della Lombardia e dell'Emilia Romagna, la genotipizzazione dei ceppi di M. bovis e M. caprae isolati nei focolai di TB delle due regioni. Inoltre il riscontro di alcune positività per M. bovis nei cinghiali della regione Marche, ha fornito l'occasione per avviare il progetto di Ricerca Corrente denominato "Epidemiologia dei micobatteri negli ungulati: indagini nell'interfaccia tra animali domestici e selvatici". La ricerca, iniziata nel 2005 e conclusa nel 2007, ha permesso di isolare e genotipizzare anche i ceppi di M. bovis provenienti dai cinghiali ed eseguire un confronto con i profili genetici degli isolati nei bovini provenienti dallo stesso territorio.
Materiali e Metodi
L'esame colturale per l'isolamento dei micobatteri è stato eseguito secondo le metodiche tradizionali previste dal D.M. 592/95, su terreni solidi selettivi (Lowestein-Jensen e Stonebrink).
L'eventuale crescita delle colonie è stata monitorata settimanalmente per un periodo di almeno tre mesi. Sulle colonie tipiche, risultate positive alla colorazione per acido resistenti, l'identificazione è stata eseguita mediante tecniche biomolecolari. Per una prima differenziazione tra Mycobacterium spp., Mycobacterium avium e Mycobacterium tuberculosis complex dalle colonie isolate è stata eseguita una metodica PCR secondo quanto riportato da Kulski et al., 1995.
Le colonie classificate come appartenenti al M. tuberculosis complex sono state successivamente tipizzate applicando il protocollo di Huard et al., 2003. I ceppi identificati come M. bovis sono stati caratterizzati presso il Centro di Referenza Nazionale per la tubercolosi bovina da M. bovis (TBCentro-IZSLER), con l'ausilio di tecniche di tipizzazione molecolare: Spoligotyping e analisi dei loci ETRs. Lo Spoligotyping è un saggio basato sul polimorfismo del locus DR (short ripetitive direct repeats) presente nel genoma dei micobatteri appartenenti al Mycobacterium tuberculosis complex e quindi anche a M. bovis e M. caprae.
La tipizzazione ETRs si basa sulla presenza di 5 loci (ETR da A ad E) ognuno dei quali contiene un'unica sequenza ripetuta a tandem, la cui dimensione varia da 53 bp a 79 bp. Ogni ceppo di M. bovis è caratterizzato da un numero variabile di sequenze ripetute. Al fine di ottenere informazioni di ordine epidemiologico, i profili genetici dei ceppi in esame sono stati confrontati con ceppi isolati nello stesso arco di tempo, nelle regioni Umbria e Marche e nel territorio nazionale.
I dati relativi alle movimentazioni degli animali ed all'identificazione degli allevamenti correlati, sono stati estrapolati dalle "Schede di rilevamento dati in allevamento" redatte dai colleghi del Servizio Veterinario nel corso dell'indagine epidemiologica nei focolai.
Risultati
I 61 ceppi di M. bovis e M. caprae tipizzati (50 isolati nei focolai bovini delle due regioni e 11 isolati nei cinghiali delle Marche) sono stati differenziati in 8 spoligotipi diversi, comprendenti M. bovis SB0120, diffuso in Umbria e Marche e M. caprae, presente solo in Umbria (Figure 1 e 2). Gli 8 spoligotipi sono stati ulteriormente differenziati con il saggio ETRs in 21 sottogruppi (Tabelle 1,2,3).
Tra questi sono stati individuati 4 cluster genetici in cui è stato possibile risalire ai collegamenti tra i diversi focolai. Il Mycobacterium bovis SB0120, caratterizzato dal profilo ETR 33533, è un cluster che è comparso tra il 2004 e il 2006 in una zona del maceratese, nelle immediate vicinanze del Parco dei Sibillini, in bovini e cinghiali che condividevano il pascolo (Focolaio 21 e C1).
In particolare nel caso dei bovini si trattava di circa 80 capi, mantenuti allo stato brado, nei quali il riscontro di ripetute positività sia in vivo che post mortem ha comportato l'abbattimento dell'intera mandria nel 2005. Per quanto riguarda i cinghiali, i capi risultati positivi all'isolamento provenivano dalle battute di caccia del 2004, 2005 e 2006, effettuate nello stesso territorio dove pascolavano i bovini. Nel caso del M. caprae, lo spoligotipo SB0418, isolato in Umbria nel 2006 e 2007, è stato evidenziato anche in altre regioni italiane. L'indagine epidemiologica, condotta nei focolai umbri, ha però consentito di ricostruire le movimentazioni degli animali e di individuare una stalla di sosta umbra (Focolaio 16) che acquistava vitelli da aree del Sud Italia dove sono stati isolati ceppi con lo stesso genotipo.
In particolare in queste aree è stato isolato SB0418 combinato con gli ETRs 63525, 53535 e 53525, genotipi isolati anche nelle aziende umbre che nel 2007 acquistavano dalla stalla di sosta (Focolaio 14, 15). Inoltre nel 2006 da capi provenienti da un allevamento da ingrasso (Focolaio 9), apparentemente non correlato con le aziende sede dei focolai del 2007, è stato isolato un M.caprae SB0418/ETR53525; anche in questo caso, grazie ad un'attenta analisi sull'origine dei capi, si è riusciti a risalire alle stesse aziende del Sud Italia da cui la stalla di sosta umbra acquistava nel 2007 e quindi è stato possibile correlare tutti i focolai.
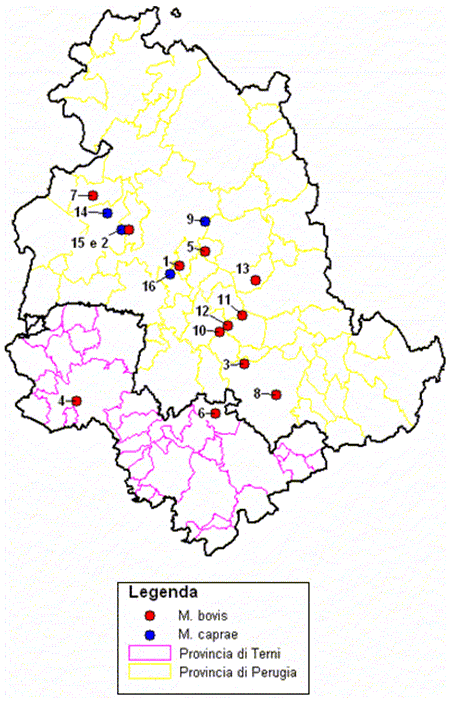
Figura 1. Localizzazione geografica dei Focolai di TB nella regione Umbria
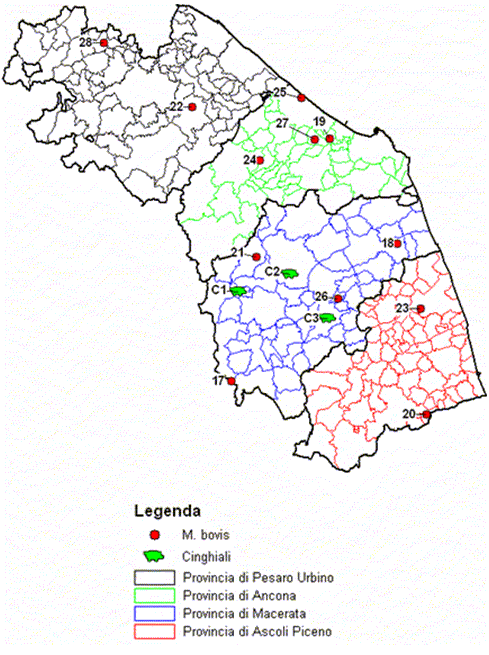
Figura 2. Localizzazione geografica dei Focolai di TB nella regione Marche
| Tabella 1. Profili genetici di M. bovis e M. caprae differenziati mediante Spoligotyping ed analisi dei loci ETRs |
| Spoligotipo |
ETRs |
Specie Animale |
Comune |
Anno |
Focolaio |
| SB0120 M. bovis |
33533 |
Bovini |
Matelica (MC) |
2004/05 |
Focolaio 21 |
| 33533 |
Cinghiali |
Matelica (MC) |
2004/05/06 |
Focolaio C1 |
| SB0120 M. bovis |
35533 |
Bovini |
Orvieto (TR) |
2003/04 |
Focolaio 4 |
| 35534 |
Bovini |
Bastia Umbra (PG) |
2005 |
Focolaio 5 |
| SB0120 M. bovis |
42533 |
Bovini |
Passignano sul Trasimeno (PG) |
2005 |
Focolaio 7 |
| 45533 |
Bovini |
Spoleto (PG) |
2005 |
Focolaio 8 |
| 45533 |
Bovini |
Spello (PG) |
2007 |
Focolaio 13 |
| 45533 |
Bovini |
San Vito (AN) |
2007 |
Focolaio 27 |
| 44533 |
Bovini |
Montefalco (PG) |
2007 |
Focolaio 11 |
| SB0120 M. bovis |
55533 |
Bovini |
Spoleto (PG) |
2003 |
Focolaio 3 |
| 54533 |
Bovini |
Spoleto (PG) |
2003 |
Focolaio 3 |
| 54533 |
Bovini |
Gualdo Cattaneo (PG) |
2007 |
Focolaio 12 |
| 54533 |
Bovini |
Visso (MC) |
2002 |
Focolaio 17 |
| 54533 |
Bovini |
Maltignano (AP) |
2003/05 |
Focolaio 20 |
| 54533 |
Bovini |
Serra de Conti (AN) |
2006 |
Focolaio 24 |
| 53533 |
Bovini |
Senigallia (AN) |
2007 |
Focolaio25 |
| 62532 |
Bovini |
Torgiano |
2002 |
Focolaio 1 |
| Tabella 2. Profili genetici di M. bovis e M. caprae differenziati mediante Spoligotyping ed analisi dei loci ETRs |
| Spoligotipo |
ETRs |
Specie Animale |
Comune |
Anno |
Focolaio |
| SB 0841 M. bovis |
35533 |
Bovini |
Acquasparta (TR) |
2005 |
Focolaio 6 |
| 52533 |
Cinghiali |
San Severino (MC) |
2005 |
Focolaio C2 |
| 52533 |
Cinghiali |
San Ginesio (MC) |
2006 |
Focolaio C3 |
| 55533 |
Bovini |
Fossombrone (PU) |
2006 |
Focolaio 22 |
| 55533 |
Bovini |
Fermo (AP) |
2006/07 |
Focolaio 23 |
| 55533 |
Bovini |
Colmurano (MC) |
2005/07 |
Focolaio 26 |
| SB 0948 M. bovis |
35533 |
Bovini |
Morrovalle (MC) |
2002 |
Focolaio 18 |
| 35533 |
Bovini |
Giano dell'Umbria (PG) |
2006 |
Focolaio 10 |
| SB 0133 M. bovis |
55533 |
Bovini |
Fossombrone (PU) |
2006 |
Focolaio 22 |
| SB 0872 M. bovis |
44533 |
Bovini |
Corciano (PG) |
2003/07 |
Focolaio 2 |
| SB 0828 M. bovis |
54533 |
Bovini |
Montegrimano (PU) |
2007 |
Focolaio 28 |
| SB 0919 M. bovis |
53553 |
Bovini |
Chiaravalle (AN) |
2003 |
Focolaio 19 |
| Tabella 3. Profili genetici di M. bovis e M. caprae differenziati mediante Spoligotyping ed analisi dei loci ETRs |
| Spoligotipo |
ETRs |
Specie Animale |
Comune |
Anno |
Focolaio |
| SB0418 M. caprae |
53525 |
Bovini |
Perugia (PG) |
2006 |
Focolaio 9 |
| 53535 |
Bovini |
Magione (PG) |
2007 |
Focolaio 14 |
| 53635 |
Bovini |
Castelvieto (PG) |
2007 |
Focolaio 15 |
| 63525 |
Bovini |
Deruta (PG) |
2007 |
Focolaio 16 |
Discussione
Come risulta anche dalla letteratura lo Spoligotyping è una tecnica scarsamente discriminante quando in una regione predominano gli stessi spoligotipi ed è stato quindi determinante affiancare l'analisi dei loci ETRs per svelare interessanti omologie tra i ceppi circolanti. Nel caso del genotipo SB120/ETR 33533, riscontrato nel maceratese, l'isolamento dello stesso ceppo di M.bovis sia dai bovini che dai cinghiali, ci ha permesso di concludere che vi è stata una circolazione del micobatterio tra le due popolazioni.
A tale riguardo resta ancora aperta la questione sul ruolo che hanno giocato gli ungulati selvatici nel contesto epidemiologico considerato, in quanto non è stato ancora chiarito se il cinghiale abbia rappresentato uno "spillover host" o un vero e proprio "resevoir". Nei focolai umbri riconducibili a M. caprae SB0418/ETRs 63525, 53535 e 53525, la ricostruzione della movimentazione degli animali ha permesso d'individuare un'area geografica dell'Italia meridionale, come possibile zona di origine dell'infezione, le informazioni ottenute dalla caratterizzazione genetica dei ceppi hanno permesso di risalire allo stesso territorio e pertanto le indagini biomolecolari hanno avvalorato le ipotesi delle indagini convenzionali.
Ad oggi non si è riusciti ad applicare questo tipo di approccio a tutti i cluster genetici individuati nel territorio umbro e marchigiano; tale osservazione induce ad approfondire ulteriormente l'indagine epidemiologica tradizionale (movimentazioni di animali, contiguità tra allevamenti, pascoli comuni) per chiarire le modalità di diffusione della malattia nel territorio considerato.
Bibliografia
Aranaz, A., Cousins, D., Mateos, A. and Lucas Domìnguez, L. (2003) Elevation of Mycobacterium tuberculosis subsp. caprae
Aranaz et al. 1999 to species rank as Mycobacterium caprae comb. nov., sp. nov. Int J Syst Evol Microbiol 53, 1785-1789.
Kulski J.K., Khinsoe C., Pryce T. and Crhistiansen K. (1995) Use of a Multiplex PCR to Detect and Identify Mycobacterium avium and Mycobacterium intracellulare in Blood Culture Fluids of AIDS Patients. J.Clin. Microbiol., Mar.1995, V.33, No3, 668-674.
Huard R.C., de Oliveira Lazzarini L.C., Butler W.R., van Soolingen D. and Ho J.L. PCR-Based Method To Differentiate the Subspecies of the Mycobacterium tuberculosis Complex on the Basis of Genomic Deletion. (2003) J. Clin. Microbiol., Apr. 2003, Vol. 41, No 4, p. 1637-1650.
Allix C., Walravens K., Saegerman C., Godfroid J., Supply P., and Fauville-Dufaux M. (2006 )Evaluation of the Epidemiological Relevance of Variable-Number Tandem-Repeat Genotyping of Mycobacterium bovis and Comparison of the Method with IS6110 Restriction Fragment Length Polymorphism Analysis and Spoligotyping J. Clin. Microbiol. 44: 1951-1962.
Aranaz, A., E. Liebana, A. Mateos, L. Dominguez, D. Vidal, M. Domingo, O. Gonzalez, E. F. Rodriguez Ferri, A. E. Bunschoten, J. D. van Embden, and D. Cousins. (1996) Spacer oligonucleotide typing of Mycobacterium bovis strains from cattle and other animals: a tool for studying epidemiology of tuberculosis. J. Clin. Microbiol. 34:2734-2740.
Haddad N.,Masselot M., Durand B. (2004) Molecular differentiation of Mycobacterium bovis isolates. Review of main techniques and applications Res Vet Sci. Feb;76(1):1-18. Review.
Roring, S., Scott A., Brittain D., Walker I., Hewinson G., Neill S., and Skuce R. A.. (2002) Development of variable-number tandem repeat typing of Mycobacterium bovis: comparison of results with those obtained by using existing exact tandem repeats and spoligotyping. J. Clin. Microbiol. 40:2126-2133.
Boniotti, M.B., Goria, M., Loda, D., Garrone, A., Benedetto, A., Mondo, A., Tisato, E., Zanoni, M., Zoppi, S., Dondo, A., Tagliabue, S., Bonora, S., Zanardi,G. and Pacciarini M.L. (2009) Molecular Typing of Mycobacterium bovis Strains Isolated in Italy from 2000 to 2006 and Evaluation of Variable-Number Tandem Repeats for Geographically Optimized Genotyping . J Clin Microbiol 47, 636-644.
Affiliazione:
Mazzone P. (a), Boniotti M.B.(b), Biagetti M.(a), Crotti S. (a), Ciullo M. (a), Cagiola M. (a), Corneli S. (a), Duranti A. (a), Faccenda L. (a), Gavaudan S. (a), Pacciarini M.L.(b)
(a) Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
(b) Istituto Zooprofilattico Sperimentale della Lombardia e dell'Emilia Romagna, Brescia - Centro di Referenza Nazionale per la Tubercolosi bovina da M. bovis



