Biblioteca Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Sanità Pubblica Veterinaria: Numero 73, Agosto 2012 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#556
Studio collaborativo per la validazione di una metodo analitico in Real Time PCR per la quantificazione di ingredienti di specie vegetali in matrici complesse - Report of a collaborative study for the validation of Real Time PCR method for the quantification of vegetable ingredients in complex matrices like feeds
Ciarrocchi F., Trozzi C., Gatto F., Briscolini S., Blasi G.
Abstract. A collaborative study has been carried out with the aim of developing and validating a Real Time PCR method for the quantification of soybean in complex vegetal matrices like feeds. The collaborative study involved eight laboratories selected among the most experienced in the application of molecular analysis on food and feed materials.
All laboratories received blind samples, reagents and a detailed protocol for reaction mixture preparation and plate set up. Blind samples were delivered as DNA solutions containing 1.5 and 5 % of soy.
Parameters such as precision, sensibility and PCR efficiency were evaluated.
The overall results were satisfactory, with a good level of reproducibility and a high sensibility. As a preliminary study, the research demonstrated that quantitative Real Time PCR assays could be employed to monitor the labeling system of a single ingredient in complex vegetable foods. Many factors however, such as composition, technology applied for feed production, quantity and purity of the extracted DNA could affect the quantification of the ingredient, then further studies are recommended in order to evaluate these parameters
Riassunto. In questo articolo sono presentati i risultati di uno studio collaborativo finalizzato alla validazione di un metodo analitico in Real Time PCR per la quantificazione relativa del contenuto di soia (Glycine max) in campioni costituiti da più ingredienti. Lo studio ha coinvolto otto laboratori che hanno eseguito la quantificazione, mediante Real Time PCR, della soia presente in due campioni appositamente preparati, contenenti rispettivamente il 5 e l'1.5 % di soia. Il metodo impiegato ha permesso di ottenere risultati soddisfacenti presentando un buon livello di riproducibilità ed una elevata sensibilità.
I risultati ottenuti dimostrano la validità del metodo come studio preliminare per la quantificazione dell'ingrediente soia in mangimi e alimenti e suggeriscono l'impiego di un simile approccio analitico per ottenere dati quantitativi sul livello di contaminazione delle filiere da parte di specie vegetali diverse da quelle normalmente impiegate
Introduzione
Per mangimi o alimenti geneticamente modificati si intendono prodotti che contengono, sono costituiti o prodotti a partire da Organismi Geneticamente Modificati (OGM). L'eventuale presenza di OGM negli alimenti, nei mangimi e in generale nella catena agroalimentare, costituisce motivo di grande interesse di tipo sanitario, ambientale, etico, socio-economico e politico per gli enti preposti ai controlli e alla certificazione, responsabili della rispondenza dei prodotti agroalimentari a standard di qualità. A tal fine, la normativa vigente in materia di autorizzazione, etichettatura e tracciabilità degli alimenti e mangimi costituiti o derivati da OGM (Regolamenti CE n. 1829/2003 e 1830/2003) fissa la soglia di contaminazione accidentale o tecnicamente inevitabile allo 0.9% di OGM rispetto al singolo ingrediente.
La Direttiva 96/25/CE stabilisce una tolleranza del 5% di impurità botaniche nelle materie prime utilizzate per la produzione di mangimi, al di sotto di questa soglia non è obbligatorio indicare la loro presenza in etichetta.
L'utilizzo di tecniche molecolari per il controllo di OGM in mangimi ha portato a rilevare la presenza di questi ingredienti a volte di derivazione GM e ha posto quindi la necessità di valutare se la presenza di tali impurità sia riconducibile ad una contaminazione accidentale, e quindi al di sotto del 5%, oppure un errore di etichettatura.
Il presente lavoro si è posto come obiettivo quello di sviluppare uno strumento analitico che consentisse di valutare in maniera più accurata possibile il contenuto dell'ingrediente soia in matrici complesse in maniera da valutare la corrispondenza dell'etichettatura.
In particolare è stata validata una metodica Real Time PCR in grado di quantificare, in campioni costituiti da più ingredienti, la percentuale della singola specie vegetale presente con particolare riferimento all'ingrediente soia.
Lo studio collaborativo ha coinvolto otto laboratori ed ha permesso di eseguire la quantificazione della soia presente in due campioni appositamente preparati.
Materiali e Metodi
Sono stati coinvolti nove laboratori (LAB 1-9): i materiali ed i campioni contenenti combinazioni fisse di materie prime e quantità di soia a percentuale variabile sono stati preparati dal Lab 1 e distribuiti ad otto laboratori partecipanti allo studio collaborativo (LAB 2-9).
I campioni sono stati formulati da DNA estratto da ciascuna matrice di mais, crusca, girasole e soia. Per ciascuna specie il DNA genomico totale è stato estratto secondo quanto riportato nella Norma ISO 21571:2005 - Annex A.3 e B.1-B.2.
Dalla massa, completamente omogeneizzata mediante omogeneizzatore Osterizer 6805-50 (Steroglass, Perugia, Italia), sono stati allestiti due sottocampioni. Nel caso della soia non si è proceduto alla predisposizione dei sottocampioni.
Da ciascun sottocampione e dal campione di soia sono stati prelevati 2 g di materiale per l'estrazione del DNA mediante trattamento chimico ed enzimatico (CTAB precipitante) e sua purificazione con kit commerciale (metodo CTAB/QIAGEN). Il DNA di tutti gli estratti è stato quantificato mediante analisi fotometrica e, ove necessario, diluito alla concentrazione di 30 ng/µL. Gli estratti così ottenuti sono stati riuniti in un'unica provetta, differente per ciascuna materia prima.
Il DNA estratto è stato impiegato per l'allestimento di due campioni (Campione A e Campione B), ciascuno costituito da:
- DNA eterologo: composto da 50 % di mais, 33 % di crusca, 17 % di girasole;
- DNA di soia: 5 % nel Campione A e 1,5% nel Campione B.7
La specifica formulazione del DNA eterologo è stata scelta in base ai risultati di un precedente studio in cui sono state ottimizzate le condizioni di amplificazione ed è stata valutata l'influenza della quantità della singola materia prima all'interno della matrice complessa.
Per ciascun campione sono state preparate due provette (A1 e A2; B1 e B2) per permettere ai laboratori partecipanti al circuito di esprimere il risultato come media di due valori, in analogia a quanto effettuato normalmente nella routine diagnostica.
È stato inoltre preparato un ulteriore campione (Campione C) costituito da DNA Soia alla concentrazione del LOD (Limite di determinazione) del metodo definita in una precedente fase di validazione (0.1 %) (Ciarrocchi et al., 2012).
Per i materiali di riferimento (calibratori) è stata impiegata la formulazione della Curva A, già allestita e saggiata in un precedente lavoro di validazione (Ciarrocchi et al., 2012). In particolare sono stati predisposti 4 preparati, costituiti da DNA eterologo secondo le concentrazioni sopra riportate e da DNA di soia nelle concentrazioni rispettivamente di 50 %, 10 %, 5 %, 1 %. Contestualmente ai campioni e ai calibratori, sono stati preparati anche i reagenti per la Real time PCR.
A ciascun laboratorio che ha partecipato allo studio (Tabella 1) è stata inviata la documentazione di accompagnamento con le indicazioni del contenuto del pacco inviato, il protocollo di analisi da seguire ed un file in formato Excel per l'inserimento dei risultati ottenuti, contestualmente al materiale, conservato in ghiaccio secco, allestito secondo quanto segue:
- PROVETTA CALIBRATORI: n. 4 provette contenenti ciascuna 18 µL di DNA da utilizzare per i quattro punti della Curva di calibrazione (Soia 1 %, 5 %, 10 %, 50 %);
- PROVETTA CAMPIONE A: n. 1 provetta "A1" contenente 18 µL di DNA e n. 1 provetta "A2" contenente 18 µL di DNA;
- PROVETTA CAMPIONE B: n. 1 provetta "B1" contenente 18 µL di DNA e n. 1 provetta "B2" contenente 18 µL di DNA;
- PROVETTA CAMPIONE C: n. 1 provetta "C" contenente 18 µL di DNA Soia alla concentrazione del LOD (0.1 %);
- PROVETTA REAGENTI: n. 1 provetta contenente 120 µL di Acqua Nuclease-free per PCR (ddH2O); n. 2 provette contenenti 90 µL rispettivamente dei primer GmaxF (10 µM) (5' - CTT TCT CGC ACC AAT TGA CA - 3') e GmaxR (10 µM) (5' - TCA AAC TCA ACA GCG ACG AC - 3'); n. 1 provetta contenente 460 µL di TaqMan Universal Master Mix (2X) (Applied Biosystems, Foster City, CA); n. 1 provetta contenente 90 µL di Sonda Gmax (5 µM) (5'-Vic - CCA CAA ACA CAT GCA GGT TAT CTT GG - Tamra - 3') (Ciarrocchi et al., 2012).

Ciascun laboratorio coinvolto nello studio collaborativo ha condotto la Real Time PCR per il sistema endogeno (lectina) impiegando i materiali di riferimento, i reagenti ed i campioni ricevuti. Dei campioni A e B non è stata rivelata la concentrazione.
La reazione è stata condotta allestendo una miscela di reazione di 25 µL contenente: TaqMan Universal Master Mix 1X; 0.9 µM di ciascun primer; DNA templato 5 µL, e impostando il seguente ciclo termico: preattivazione UNG (Uracil-N-glicosilasi) a 50 °C per 2 minuti; preattivazione DNA polimerasi e denaturazione DNA a 95 °C, 10 minuti; 45 cicli di denaturazione a 95 °C per 15 secondi, annealing e sintesi a 60 °C per 60 secondi.
Ciascun punto delle curve di calibrazione e ciascun campione sono stati analizzati in un'unica sessione di PCR e in triplicato per ottenere una migliore affidabilità nel calcolo del ciclo soglia. Come controllo di amplificazione ogni laboratorio ha impiegato un proprio controllo contenente soia. La linearità e l'efficienza delle curve di calibrazione sono state valutate come descritto da Ciarrocchi e collaboratori (2012).
La costruzione della retta di calibrazione ha permesso a ciascun laboratorio di quantificare (%) la soia presente nei campioni A e B. A tale scopo, sono stati interpolati i corrispondenti valori di Ct ottenuti da ciascun estratto e ne è stata calcolata la media della corrispondente quantità di soia presente.
Al fine di valutare la concordanza dei risultati e di individuare eventuali differenze statisticamente significative tra i diversi laboratori coinvolti nello studio collaborativo (P < 0.05), i risultati delle medie delle quantità di soia presenti nei campioni, espresse in percentuale, sono stati sottoposti ad analisi della varianza ad una via (ANOVA) utilizzando il software JMP (versione 3.1.5).
La validazione del metodo è stata eseguita valutando la sensibilità, in accordo con quanto stabilito dalle linee guida adottate per la valutazione di metodi di Real Time PCR per la quantificazione di OGM (MPR ENGL http://gmo-crl.jrc.ec.europa.eu/)
Risultati
I risultati dello studio collaborativo sono stati organizzati, valutati ed elaborati dal LAB 1 in collaborazione con il LAB 3 ed il LAB 4.
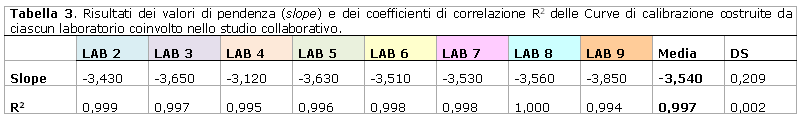
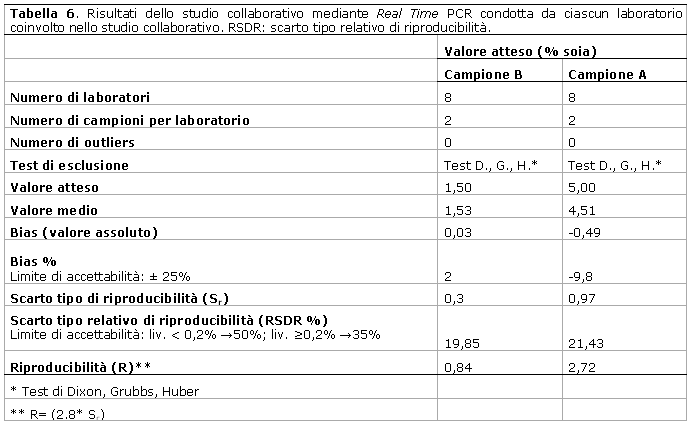
In Tabella 2 sono riportati i risultati dei saggi in Real Time PCR condotti sui quattro punti della curva di calibrazione costruita da ciascun laboratorio coinvolto nello studio collaborativo, mentre in Tabella 3 vengono elencati i risultati dei valori di pendenza (slope) e del coefficiente di correlazione R2 relativi alla stessa curva.
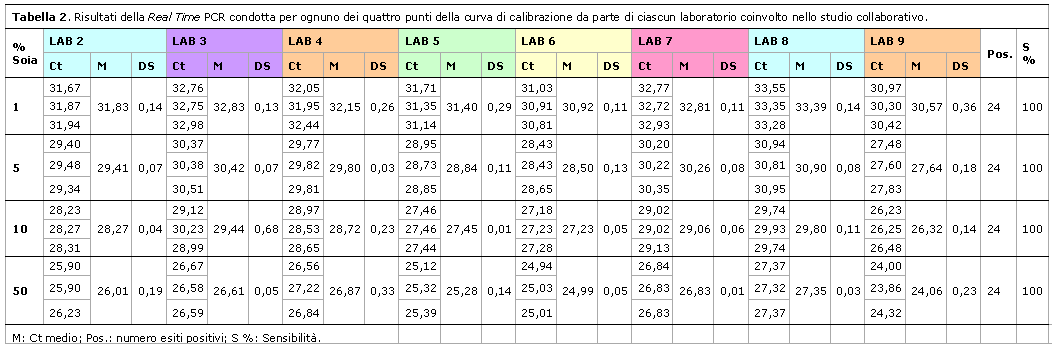
La linearità della reazione del sistema endogeno è stata osservata in tutti i laboratori coinvolti. Infatti, tutte le curve hanno raggiunto un valore accettabile del coefficiente di correlazione R2, con valori sempre superiori o uguali a 0.98. Anche il valore medio dei coefficienti R2 è rientrato nei criteri di accettabilità, raggiungendo il valore di 0.997. La linearità è stata inoltre confermata dalla diminuzione dei valori medi di Ct, contestualmente all'aumento della concentrazione del DNA di soia nei materiali di riferimento.
I laboratori non hanno ottenuto la medesima efficienza di amplificazione delle curve, espressa dai valori di slope, che tuttavia sono risultati accettabili e compresi tra -3.1 e -3.6, ad eccezione dei LAB 3 e 9 per i quali la minore efficienza di amplificazione è stata rivelata da valori inferiori di slope rispettivamente di -3.65 e di -3.85.
Il valore medio di slope è stato di -3.54 e pertanto conforme ai criteri di accettabilità delle curve di calibrazione, con una efficienza di amplificazione del 100 %.
In Tabella 4 sono elencati i dati grezzi ottenuti dai saggi in Real Time PCR condotti sui campioni A e B, sul campione C e sui controlli di amplificazione.
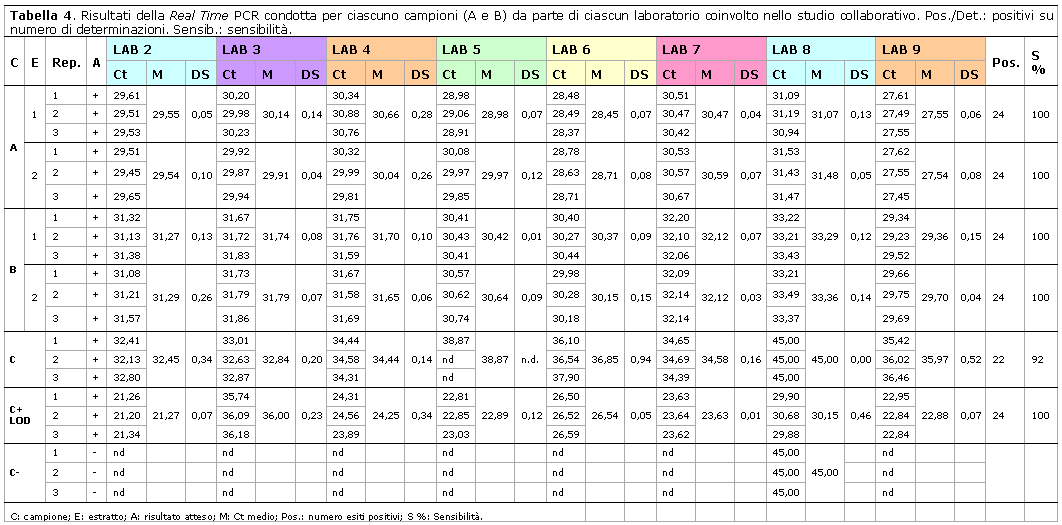
Sebbene i risultati ottenuti abbiano mostrato una sensibilità della reazione pari al 100 % per i campioni A e B, non è stato possibile confrontare i valori di Ct registrati dai diversi laboratori coinvolti poiché ottenuti impostando valori di threshold differenti.
Il campione C, preparato alla concentrazione del LOD del metodo (0.1 %), è stato analizzato da ciascun laboratorio per la valutazione della sensibilità del metodo, che è risultata del 92 %.
La quantificazione della soia presente nei campioni A e B è stata condotta da ogni laboratorio mediante la retta di calibrazione, interpolando il valore del Ct ottenuto per ciascun estratto, e calcolando la media dei valori ottenuti.
La Tabella 5 riporta i risultati delle prove di quantificazione della soia presente nei campioni A e B, ottenuti presso i laboratori coinvolti nello studio collaborativo. Per ciascun campione è riportato il valore medio della percentuale di soia, calcolato sulla media di due misure di quantificazione (ciascuna proveniente da due estratti analizzati in Real Time PCR).
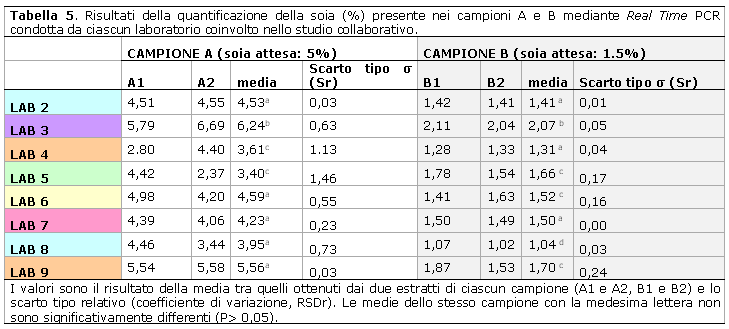
I laboratori che hanno ottenuto quantificazioni di soia più vicine ai valori attesi sono stati LAB 6 per entrambi i campioni, LAB 2 e LAB 7 rispettivamente per il campione A e per il campione B.
Al fine di valutare la concordanza dei risultati di tutti i laboratori coinvolti nello studio collaborativo, è stata condotta l'analisi ANOVA sulle medie delle percentuali di quantificazione ottenute dai laboratori (Tabella 5). L'inferenza sulle differenze significative tra le medie ha permesso di formulare differenti considerazioni a seconda del campione analizzato:
- Campione A (valore atteso 5 %): nella maggior parte dei laboratori (63 %) la percentuale media di soia quantificata non è stata significativamente differente (P > 0.05), raggiungendo valori compresi tra un massimo di 5.56 per LAB 9 e 3.95 per LAB 8. I valori minimi raggiunti e registrati dai LAB 4 e 5, con valori percentuali rispettivamente di 3.61 e 3.40, sono risultati significativamente differenti (P < 0.05) a quelli ottenuti dalla maggior parte dei laboratori e a quello ottenuto dalla LAB 3 con il maggiore quantitativo di soia rilevata pari al 6.24 %.
- Campione B (valore atteso 1.5 %): è stata osservata una maggiore variabilità tra le percentuali medie ottenute dai laboratori. Il valore maggiore è stato ottenuto da LAB 3, con il 2.07 % di soia quantificata, risultato significativamente differente da tutti gli altri valori (P < 0.05). Inoltre, è stata osservata una differenza significativa anche tra il valore più basso, registrato da LAB 8 con il 1.04 % di soia, ed il resto dei dati ottenuti dagli altri laboratori.
I risultati ottenuti sono stati quindi elaborati per calcolare lo scarto tipo di riproducibilità e la riproducibilità del metodo impiegato che è risultata di 2.72 nel caso del campione A e di 0.84 nel campione B (Tabella 6).
Discussione
I risultati dello studio collaborativo presentati in questo lavoro hanno visto il coinvolgimento di otto laboratori che hanno condotto i saggi in Real-time PCR per la quantificazione della soia presente in due campioni, A e B, alle concentrazioni rispettivamente di 5 % e di 1.5 %.
L'analisi statistica effettuata dimostra che non vi è sempre una concordanza ottimale tra i risultati della quantificazione ottenuti dai laboratori, in entrambi i campioni analizzati. Va però anche segnalato che per il campione A, a maggior concentrazione di soia, si sono ottenuti risultati più affini tra i diversi laboratori. Questi dati suggeriscono un miglioramento della riproducibilità del metodo all'aumentare della concentrazione di DNA bersaglio presente nel campione.
I risultati raggiunti hanno permesso di osservare nel complesso un buon livello di riproducibilità del metodo impiegato. Tuttavia, la concordanza non sempre elevata tra i risultati espressi dai diversi laboratori, può essere dovuta a diversi fattori quali i materiali di riferimento utilizzati, l'omogeneità dei campioni preparati, la diversa manualità degli operatori coinvolti nelle analisi, la composizione ed i trattamenti tecnologici associati alle diverse fasi di produzione delle matrici analizzate.
La normativa vigente stabilisce che non debba essere sottoposto ad etichettatura una materia prima che sia presente in quantità inferiore al 5 % mentre la soglia di contaminazione accidentale o tecnicamente inevitabile di OGM (0.9 %) è riferita ai singoli ingredienti indipendentemente dalla quantità presente in alimenti complessi.
La composizione ed i trattamenti tecnologici associati alla preparazione dei mangimi rendono a volte difficile valutare l'accidentalità della presenza di specie vegetali non dichiarate. Oltretutto, è ancora controversa la definizione di opportuni materiali di riferimento. Il presente lavoro rappresenta un possibile approccio preliminare alla risoluzione del problema della quantificazione dell'ingrediente soia nei mangimi costituiti da matrici eterogenee.
La sensibilità e l'accuratezza osservate consentono di ipotizzare l'impiego dello stesso approccio analitico per la valutazione delle impurità botaniche nei piani di autocontrollo aziendali.
Ringraziamenti
Lo studio è stato finanziato dal Ministero della Salute e fa parte del progetto di ricerca corrente dal titolo "Messa a punto e validazione di una metodo analitico in Real Time PCR per la quantificazione di ingredienti (specie vegetali) in matrici complesse" (RC IZSUM 20/2008). Si ringraziano tutti i laboratori che hanno partecipato allo studio collaborativo: Istituto Zooprofilattico Sperimentale Umbria e Marche, Istituto Zooprofilattico Sperimentale Lazio e Toscana, Istituto Zooprofilattico Sperimentale della Sardegna, Istituto Zooprofilattico Sperimentale della Lombardia e dell' Emilia Romagna "Bruno Ubertini", Istituto Zooprofilattico Sperimentale del Mezzogiorno, Istituto Zooprofilattico Sperimentale della Sicilia "Adelmo Mirri", Istituto Zooprofilattico Sperimentale del Piemonte, della Liguria e della Valle D' Aosta, Bioaesis s.r.l.
Bibliografia
Berdal, KG., Holst-Jensen A. (2001). Roundup Ready® soybean event-specific real-time quantitative PCR assay and estimation of the practical detection and quantification limits in GMO analyses. European Food Research and Technology, 213: 432-438.
Ciarrocchi F., Canonico C., Trozzi C., Gatto F., Briscolini S., Blasi G. (2012). Validazione di un metodo analitico in Real Time PCR per la quantificazione di ingredienti di specie vegetali in matrici complesse: studio preliminare. Sanità Pubblica Veterinaria, Numero 71, Aprile 2012 [http://spvet.it/] ISSN 1592-1581.
DIRETTIVA 95/25/CE - circolazione e utilizzazione delle materie prime per mangimi, che modifica le direttive 70/524/CEE, 74/63/CEE, 82/471/CEE, 93/74/CEE e che abroga della direttiva 77/101/CEE.
European Network of GMO Laboratories (ENGL). Definition of Minimun Performance Requirements for Analytical Methods of GMO Testing, (13 October 2008).
Martinelli, L, Benvenuto, E, Dalla Costa, L, Nobili, C (2007). Manuale di laboratorio per l'analisi di OGM in matrici agroalimentari. ENEA Ente per le Nuove tecnologie l'Energia e l'Ambiente. Roma.
REGOLAMENTO (EC) 1829/2003 sull'immissione sul mercato di alimenti e mangimi contenenti, costituiti o derivati da OGM.
REGOLAMENTO (EC) 1830/2003 sulla tracciabilità ed etichettatura di OGM e sulla tracciabilità di alimenti e mangimi ottenuti da OGM.

