La soia (Glycine max) è una leguminosa di elevata importanza economica e nutrizionale. Si differenzia nettamente dagli altri legumi per l'elevato tenore proteico (36,9%) e in lipidi (18,1%). Le proteine più abbondanti sono quelle di riserva localizzate nei corpi proteici, presenti in grandi quantità nelle cellule dei cotiledoni. La soia contiene nella stessa proporzione (11%): amido, glucidi solubili (esosi, saccarosio, raffinosio e altri) e fibra, oltre a discrete quantità di sali minerali (K, Fe, Ca, P, Zn) e di tiamina.
Il seme di soia è inoltre ricco in aminoacidi essenziali tra cui lisina, leucina, isoleucina e valina. Si ritrovano basse percentuali di estrogeni, gozzigeni, fitati, saponine, lectine, inibitori di proteasi, ureasi e fattori allergici. Tutti questi composti "antinutrizionali" vengono denaturati dal calore o eliminati per estrazione durante la lavorazione della soia.
La soia ha vasto uso e impiego nell'industria alimentare dalla quale vengono ricavati vari prodotti tra cui le bevande di soia (ottenute dalla farina), prese in esame in questo studio. La notevole quantità di soia presente in Italia, per una produzione nazionale scarsa, proviene da Paesi (come U.S.A., Argentina, Canada) che permettono la libera circolazione di soia geneticamente modificata, o Roundup Ready, caratterizzata dalla resistenza all'erbicida Roundup (prodotto, come la soia RR dalla multinazionale Monsanto).
Tipicamente la scelta di un erbicida si basa su vari fattori: spettro d'azione, costi e caratteristiche d'impiego.
Molti erbicidi disponibili oggi, rispondono a queste caratteristiche, ma molti sono non selettivi. Inoltre in alcuni batteri del suolo sono stati scoperti, e poi isolati e clonati, alcuni geni che codificano per enzimi capaci di inattivare per acetilazione o idrolisi gli erbicidi totali. Un erbicida non selettivo è il glifosato (N-phosphonomethyl-glycine) ingrediente attivo nell'erbicida Roundup (1) altamente attivo contro la gran parte delle infestanti.
Mediante le tecnologie d'ingegneria genetica, è stato possibile inserire nel DNA di soia un gene esogeno che rende questa specie vegetale significativamente tollerante al glifosato (2, 3, 4, 5). E' stata così prodotta la soia Roundup Ready (6), che porta come transgene un gene ibrido, EPSPS, che la rende resistente all'erbicida Roundup.
L'enzima 3-enolpiruvil-shikimat-5-fosfato-sintetetasi (EPSPS) catalizza la fissazione del fosfoenolpiruvato (PEP) sul shikimat-5-fosfato, un prodotto intermedio della sintesi del corismato, sulla cui base sono formati gli aminoacidi aromatici: fenilalanina, tirosina e triptofano.
L'EPSPS interviene al momento della biosintesi del corismato, questa via di biosintesi si trova nei batteri, nei funghi e nelle piante, ma è assente nelle cellule animali. L'EPSPS si trova nei cloroplasti o nei plastidi delle piante (7). L'erbicida glifosato (N-phosphonometilglicina, commercialmente Roundup) è un inibitore competitivo di questa tappa di sintesi (8, 9).
L'EPSPS, ottenuto dal battere Agrobacterium tumefaciens ceppo CP4, ha la caratteristica naturale di essere molto poco inibito dal glifosato, senza pertanto perdere l'affinità per il substrato naturale, cioè il Fosfoenolpiruvato, PEP (10). La struttura del frammento esogeno introdotto nel genoma della pianta, comprende il gene e delle sequenze regolatrici della sua espressione, oltre a geni marcatori che indicano l'avvenuta modificazione genica.
Il transgene della soia Roundup Ready di Monsanto, cioè il gene codificante la 3-enolpiruvil-shikimat-5-fosfato-sintetasi (EPSPS), deve essere trasportato nel DNA dei cloroplasti (7).
Questo è possibile mediante una sequenza segnale CTP (Chloroplast Transit Peptid), del gene EPSPS, ottenuto da Petunia ibrida, clonata davanti al gene CP4-EPSPS (9), ottenuto dal batterio Agrobacterium tumefaciens e responsabile della tolleranza Roundup (3, 4).
Il gene ibrido Petunia-Agrobacterium è controllato dal promotore forte CaMV 35S del virus del Mosaico del Cavolfiore, attivo in tutte le piante (11). L'espressione del transgene viene terminata mediante un segnale di poliadenilazione e un terminatore, NOS (gene della nopalina-sintetasi) ottenuto sempre dal batterio Agrobacterium tumefaciens.
LEGISLAZIONE
La soia Roundup Ready è stata autorizzata e approvata per il mercato (12). Il consenso per l'importazione nell'Unione Europea è stato preso, dopo il 1995, con la Decisione della Commissione 96/281/EC del 3 Aprile 1996.
La normativa (12) per gli alimenti ad uso umano permette l'utilizzo di Organismi Geneticamente Modificati (OGM) a condizione che sull'etichetta del prodotto sia specificamente dichiarata la presenza di ingredienti o additivi derivati da organismi geneticamente modificati.
In tal senso, il Regolamento (CE) 1139 del 26 Maggio 1998, rendeva genericamente necessario riportare tale informazione sull'etichetta (13).
Normative successive, applicabili agli alimenti per uso umano, hanno introdotto una soglia di tolleranza, intesa come livello accettabile per "contaminazione accidentale", dell'1%, valore al di sotto del quale non è previsto l'obbligo di indicare gli ingredienti transgenici presenti (Regolamento CE 49 del 10 Gennaio 2000) (14). Anche per gli additivi ed aromi geneticamente modificati (GM) o derivati da organismi GM vale l'obbligo della dichiarazione in etichetta (Regolamento CE 50 del 10 Gennaio 2000), non è però prevista alcuna soglia di tolleranza (15).
Deve essere invece garantita l'esclusione di ingredienti e/o additivi ed aromi GM negli alimenti destinati alla prima infanzia (D.P.R. 7 Aprile 1999, n° 128) e nei prodotti alimentari biologici (Decreto Ministeriale 4 Agosto 2000; G. U. n° 211 09/09/2000).
L'introduzione nel territorio Europeo, di soia transgenica ed il suo conseguente impiego nell'alimentazione umana ed animale, ha generato la necessità di controllarne la presenza in varie matrici alimentari, rinforzando l'interesse nei confronti dei metodi di rilevazione (16, 17).
La necessità di disporre di opportune metodiche analitiche è stata incrementata significativamente anche dalla presenza di miscele di distinti prodotti GM e dalla varietà di complessi processi di preparazione degli alimenti (18, 19).
Quindi, affinché sia garantito il rispetto delle normative vigenti sull'etichettatura di prodotti alimentari, è indispensabile che gli organi di controllo preposti, in particolare i laboratori degli Istituti Zooprofilattici Sperimentali, ufficialmente incaricati dal Ministero della Salute ad effettuare le determinazioni analitiche previste, dispongano di metodi sempre più specifici e sensibili, nonché, possibilmente, rapidi ed economici.
In generale qualsiasi strategia di rivelazione per risultare valida deve essere specifica, sensibile e semplice. Specifica vuol dire che il saggio in questione deve fornire una risposta positiva esclusivamente per l'organismo o la molecola bersaglio. Sensibile significa che il saggio diagnostico deve essere in grado di identificare quantità piccolissime dell'organismo o delle molecole bersaglio. La semplicità è necessaria affinché il saggio possa essere condotto efficientemente, efficacemente e a costo contenuto nella routine. La presenza di OGM negli alimenti e nei mangimi può essere determinata mediante ricerca del DNA transgenico, metodo biomolecolare basato sull'amplificazione genica (PCR) (20, 21, 22). Il grande vantaggio dei metodi basati sulla ricerca del DNA transgenico è quello di permettere l'identificazione degli OGM anche in matrici che abbiano subito trattamento termico o chimico intenso, ai quali il DNA risulta relativamente resistente.

MATERIALI E METODI Sono stati analizzati sette campioni di bevande di soia di diverse marche, di cui tre prelevati da banconi di supermarket, due dichiarati biologici acquistati in negozi specializzati e due destinati all'infanzia, acquistati in farmacia.
Le bevande di soia vengono prodotte mediante trattamenti chimici e fisici, che possono modificare considerevolmente l'ammontare e la quantità del DNA dei vari ingredienti. La ripetibilità e la riproducibilità dei metodi di estrazione può quindi variare molto da campione a campione e molto spesso i protocolli vanno adattati alla tipologia merceologica del campione (23).
Il principio basilare dell'estrazione consiste nel liberare il DNA presente nell'alimento in seguito a lisi cellulare, purificazione del DNA ottenuto da eventuali inibitori della PCR e sospensione del DNA in acqua sterile o tampone TE (Tris-EDTA).
Il DNA di ognuno dei sette campioni è stato estratto con cinque metodiche diverse: Mag-Easy (Tecna) (24), Wizard Magnetic DNA Purification System for Food (Promega), CTAB (hesadecyl-trimethyl ammonium-bromide) (25), QIAamp DNA Mini Kit (QIAGEN), Dneasy Plant Maxi Kit (QIAGEN) per valutare l'efficacia dei metodi di estrazione in relazione alla matrice.
In ogni metodica di estrazione adottata, è stato utilizzato: un controllo positivo (IRMM certified Reference Material Soya Bean Powder 2% FLUKA), un controllo negativo (IRMM certified Reference Material Soya Bean Powder 0% FLUKA), controlli negativi di estrazione (acqua ultra pura sterile invece del campione, intercalati ogni quattro provette, per verificare la potenziale contaminazione durante il processo), infine i campioni che sono stati analizzati in doppio.
VALUTAZIONE QUALI-QUANTITATIVA DEL DNA Disporre di DNA puro, privo di proteine o altri contaminanti è importante per una buona riuscita dell'analisi qualitativa mediante PCR.
La valutazione qualitativa del DNA avviene mediante elettroforesi su gel d'agarosio allo 0.8% in TBE 1X (Tris-aminometano, Acido borico, EDTA sale bisodico).
La separazione mediante gel elettroforesi si esegue a temperatura ambiente per circa un'ora; la massima forza applicata è di 5 V/cm. Gli acidi nucleici, mediante il bromuro di etidio disciolto nel gel, sono visualizzati alla luce UV (312 nm). I risultati della separazione sono conservati mediante una fotografia del gel.
Un'ulteriore valutazione quali/quantitativa del DNA è determinata dalla concentrazione di DNA nell'eluato, misurata mediante assorbanza ad una lunghezza d'onda (l) di 260 nm e 280 nm in uno spettrofotometro (Beckman) a luce UV. La lettura dell'assorbanza dovrebbe dare valori compresi tra 0.1 e 1.0 per essere accurata. Le diluizioni dei campioni sono aggiustate in modo che l'eluato contenga dai 25 ai 50 ng di DNA /ml. La purezza è determinata calcolando il rapporto dell'assorbanza a 260 nm e 280 nm; un DNA puro ha un rapporto 260/280 di 1.7-1.9.
Calcolata la concentrazione di DNA in nanogrammi, si procede all'analisi qualitativa del DNA.
ANALISI QUALITATIVA
La strategia analitica mediante la reazione a catena della polimerasi (PCR), prevede: un primo test per la rivelazione del DNA di soia mediante rivelazione del gene lectina (26); l'eventuale positività del risultato porta ad un secondo test di screening, per l'individuazione di una modificazione genetica mediante amplificazione del promotore 35S (24, 25, 27, 28). I campioni risultati positivi sono analizzati con un terzo test per l'identificazione del transgene specifico EPSPS (18, ). La presenza o l'assenza di quest'ultimo porta alla conclusione dell'analisi qualitativa.
PRIMO TEST: RIVELAZIONE DEL DNA DI SOIA
Le reazioni di amplificazione sono eseguite usando Ready-To-Go PCR Beads (Amersham Pharmacia Biotech) in Eppendorf sterili da 0.2 ml (24).
Al tubo di reazione, contenente 1.5 unità di Taq DNA polimerasi, 10 mM di Tris HCl pH 9.0, 50 mM KCl, 1.5 mM di MgCl2, 200 mM di ciascun dNTP e BSA, sono aggiunti 5 ml di DNA, 0.2 mM di ciascun primer della coppia LE 5/LE 6, 10 mM, (LE 5: 5'-TCA ACg AAA ACg AgT CTg gTg-3', LE 6: 5'-ggT ggA ggC ATC ATA ggT AAT-3', Genset Oligos) (24) e acqua MilliQ sterile per portare ad un volume finale di 25 ml.
La PCR è condotta, in un termociclatore (Gene Amp PCR System 9600, Perkin Elmer, Applied Biosystems), per 1 ciclo a 95°C, 5 minuti seguito da 40 cicli alle seguenti condizioni: denaturazione a 95°C per 30 secondi, annealing a 60°C per 30 secondi, estensione a 72°C per 60 secondi.
Alla fine gli amplificati sono mantenuti a 72°C per 5 minuti. Il prodotto di amplificazione, di 180 bp, è separato in un gel di agarosio al 2% in TBE 1X. I risultati resi visibili dalla colorazione mediante bromuro d'etidio e dalla transilluminazione UV, per comparazione con un marcatore di DNA di dimensioni note.
SECONDO TEST QUALITATIVO DI SCREENING
Si esegue per identificare la presenza di diversi tipi di organismi ingegnerizzati in un campione. Le reazioni di amplificazione sono state condotte in un primo momento utilizzando le Ready-To-Go PCR Beads (24) e la coppia di primers 35S-1/35S-2 (35S-1: 5'-gCT CCT ACA AAT gCC ATC A-3', 35S-2: 5'-gAT AgT ggg ATT gTg CgT CA-3'); successivamente questa metodica è stata sostituita con un'altra più sensibile e specifica per prodotti finiti: sono utilizzate provette sterili da 0.2 ml, si allestisce una Mastermix, contenente: 2.5 ml del buffer 10X per la polimerasi, 1.5 mM di MgCl2, 160 mM di ciascun dNTP, 0.8 U di AmpliTaq Gold Polymerase, 0.6 mM di ciascun primer p35S-cf3/p35S-cr4 (35S-cf3: 5'-CCA CgT CTT CAA AgC AAg Tgg-3', 35S-cr4 5'-TCC TCT CCA AAT gAA ATg AAC TTC C-3') e acqua MilliQ sterile (18, 29). Al tubo di reazione sono aggiunti 5ml di DNA per un volume finale di 25 ml. La PCR è condotta in un termociclatore, per 1 ciclo di 10 minuti a 95°C, seguito da 50 cicli alle seguenti condizioni: denaturazione a 95°C per 25 secondi, annealing a 62°C per 30 secondi, estensione a 72°C per 45 secondi.
Al termine gli amplificati sono mantenuti a 72°C per 7 minuti. Il prodotto di PCR, di 123 bp, è fatto correre su gel d'agarosio al 2% in TBE 1X, e visualizzato, mediante colorazione con bromuro d'etidio, su un transilluminatore, per comparazione con un marcatore di DNA di dimensioni note.

TERZO TEST QUALITATIVO SPECIFICO
Determina la presenza di un particolare organismo geneticamente modificato, è utilizzato in questo studio per l'individuazione specifica della soia Roundup Ready.
La reazione di amplificazione è eseguita mediante l'utilizzo di Ready-To-Go PCR Beads (24). Al tubo da reazione, di 0.2 ml, sono aggiunti 5 ml di DNA, 0.2 mM di ciascun primer della coppia SPA/SPC, 10 mM, (SPA: 5'-CCA CTA TCC TTC gCA AgA CCC TTC C-3', SPC: 5'-TTg TAT CCC TTg AgC CAT GTT GT-3') e acqua MilliQ sterile. La PCR è condotta in un termociclatore, alle stesse condizioni di corsa adottate per i primer LE 5/LE 6.
Il prodotto di amplificazione di 120 bp, è visualizzato su un transilluminatore, mediante corsa elettroforetica su gel d'agarosio al 2% in TBE 1X, colorato con bromuro d'etidio.
ANALISI QUANTITATIVA: REAL-TIME PCR
I campioni caratterizzati dalla presenza di OGM nel test qualitativo, sono analizzati ulteriormente, mediante una PCR quantitativa Real-Time (30, 31).
Le soluzioni dei DNA dei campioni da analizzare, sono prima dosate allo spettrofotometro e, dove necessario, diluite per ottenere una certa uniformità nelle concentrazioni.
Secondo il saggio TaqMan sono preparate le seguenti miscele di reazione: per la Lectina , MasterMix, Lectin-F, Lectin-R (10 mM), Lectin probe (5 mM), acqua MilliQ sterile; per la Roundup Ready, MasterMix, RR-F, RR-R (10 mM), RR-probe (5 mM), acqua MilliQ sterile.
Le specifiche e le performances per l'ABI Prism 7700 Sequence Detector sono basate su 50 ml di campione per ciascun pozzetto. Su una piastra a 96 pozzetti possono essere processati: 10 campioni analizzati in triplice copia, con una curva di taratura a 5 punti (0.1%, 0.5%, 1%, 2%, 5%).
Assicurandosi dell'assenza di bolle, si chiude l'Optical Plate con gli appositi Optical Caps e la piastra viene posizionata nel Sequence Detector Sample Block. In Setup view sono impostati: la piastra con lo stesso ordine in cui è stata sistemata nel sample block; il Day Layer VIC per la lectina, il Day Layer FAM per la Roundup Ready e le condizioni di amplificazione che prevedono 3 stages: stage 1 a 50°C per 2 minuti, stage 2 a 95°C per 10 minuti, stage 3: 50 cicli con denaturazione a 95°C per 15 secondi e annealing ed estensione in un unico step a 60°C per 60 secondi.
Utilizzando l'Analysis view viene azionato il Sequence Detector. La corsa ha una durata di circa 2 ore e 20 minuti. Al termine della corsa i dati vengono analizzati, esportati su Excel, dove sono ordinati ed elaborati grazie ad un programma che calcola le percentuali di OGM.
1 2 3 4 5 6
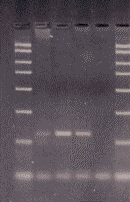
|
PCR Lectina (soia) primer LE5-LE6 (amplificato 180 bp). Marker in psizione 1 e 6; campione posizione 2; standard soia 0% posizione 3; standard soia 2% posizione 4; controllo negativo posizione 5 |
RISULTATI
Una prima verifica dei vari metodi di estrazione, adottati in questo studio, è effettuata basandosi sulla valutazione e confronto della qualità, quantità e amplificabilità dei DNA estratti.
Mediante la valutazione qualitativa del DNA, è stato verificato che dalla maggior parte delle estrazioni non si sono ottenuti DNA di buona qualità. Su gel d'agarosio è stato possibile visualizzare solamente i DNA degli standard e quelli dei campioni ottenuti con il metodo CTAB (25).
La lettura allo spettrofotometro, della quantità di DNA ottenuta, effettuata alla lunghezza d'onda (l) di 260 nm, non è stata ottenuta per i DNA di molti campioni, a causa della loro degradazione e bassa concentrazione. Nel caso di stime spettrofotometriche affidabili dei prodotti, sono state calcolate le quantità in nanogrammi dei DNA estratti.
La quantificazione mediante spettrofotometro ha dato una concentrazione media di 20 ng/ml per i DNA estratti con il metodo Mag-Easy tranne che per un campione con valori fuori range, mentre tutti i DNA estratti con il Kit QIAGEN e Wizard non hanno dato risultati validi per un'attendibile quantificazione del DNA presente nei campioni. Con il Kit Dneasy Plant i DNA estratti sono stati valutati tutti intorno agli 11 ng/ml, e tutti i DNA estratti con il CTAB sono stati ugualmente quantificabili spettrofotometricamente, ma con concentrazioni molto più alte, superiori ai 100 ng/ml.
L'amplificabilità dei DNA è valutata mediante PCR con i primers pianta specifici LE 5/ LE 6 (26, 24) che amplificano un frammento del gene lectina, specifico per la soia. Mediante quest'analisi si può confermare la presenza, la qualità e l'amplificabilità dei DNA di soia, estratti dai campioni.
Per tre campioni, risultati non amplificabili è stata verificata l'eventuale presenza di inibitori mediante una contaminazione artificiale (DNA spike), aggiungendo cioè una quantità nota di DNA modificato geneticamente al campione.
Questa ulteriore analisi, ha confermato la presenza di inibitori nei medesimi campioni, con tutti i metodi di estrazione tranne con il metodo DNeasy Plant Maxi Kit con il quale l'inibizione si è verificata solo per due dei tre campioni, e tranne con la metodica CTAB in cui tutti i campioni sono risultati amplificabili. Per diminuire l'azione inibitrice sono state ripetute le amplificazioni utilizzando DNA diluiti 1:2 e 1:10, che non hanno però dato risultati migliori, per la quantità esigua di DNA di partenza.
I campioni di controllo di estrazione sono risultati negativi, indicando assenza di contaminazioni nell'esecuzione della metodica, quindi attendibilità dei risultati ottenuti.
VERIFICA DELL'EVENTUALE PRESENZA DI OGM
I campioni, risultati amplificabili e contenenti DNA di soia, sono stati ulteriormente analizzati, per verificare l'eventuale presenza di organismi geneticamente modificati. Per conseguire tale scopo, i campioni sono stati sottoposti ad un test analitico di screening mediante PCR (24,25, 27, 28).
Questo metodo è basato sull'identificazione della più comune e frequente sequenza regolatrice, il promotore CaMV 35S, usata per alludere alla presenza di OGM, senza identificare specificatamente il transgene. L'analisi qualitativa ricorre all'utilizzo dei primer 35S-1/2 (26), che amplificano un frammento di 195 bp della sopracitata sequenza regolatrice.
Su gel d'agarosio, i controlli negativi (bianco e standard di riferimento: soia 0%) non presentavano bande, confermando la validità della reazione. Il controllo positivo (standard di riferimento: soia 2%) presentava la banda di 195 bp, mentre tutti i campioni risultavano privi di bande.
L'utilizzo di una nuova coppia di primers, 35S-cf3 e 35S-cr4 (18, 29), studiati per l'analisi di prodotti finiti, ha portato alla loro verifica sui DNA estratti dai campioni di bevande di soia. Questi primers sono posizionati nella regione corrispondente della sequenza promotrice CaMV 35S.
Al termine dell'analisi, dalla lettura su gel dei prodotti di PCR, si è potuto constatare che i controlli negativi non hanno presentato bande, quindi non è avvenuta contaminazione. Il controllo positivo ha presentato il frammento caratteristico di 123 bp. Questa banda è stata visualizzata in tutti i campioni, risultati pertanto positivi al secondo test di screening, per la rivelazione del promotore CaMV 35S.
VERIFICA DEL TRANSGENE SPECIFICO: CTP/EPSPS
Tutti i sette campioni, che hanno rivelato la presenza del promotore 35S, sono stati sottoposti ad una PCR qualitativa transgene specifica, mediante i primers SPA/SPC (24).
I primers SPA e SPC sono stati progettati e usati per l'identificazione specifica del gene CTP/EPSPS nella soia Roundup Ready. Il primer SPA è situato nella regione del promotore 35S. Il primer SPC è localizzato all'interno dell'N-terminal EPSPS Chloroplast Transit Peptide sequence (CTP).
Tutti i campioni che hanno presentato un prodotto di amplificazione di 120 bp, diagnostico per la soia geneticamente modificata, sono stati analizzati mediante PCR quantitativa, al fine di determinare in percentuale l'ammontare di transgene presente.
PERCENTUALE DI SOIA ROUNDUP READY
Lo strumento ABI PRISM 7700 Sequence Detector (Applied Biosystems), permette di rilevare quantitativamente la presenza di acidi nucleici, determinando l'esatta percentuale di costituenti geneticamente modificati di un campione, in Real Time PCR.
La tecnologia della PCR quantitativa Real Time, è legata alla fluorescenza (32, 33). Prevede l'uso di sonde ad ibridazione, TaqMan, legate a molecole fluorescenti: "reporter" all'estremita 5' e " quencher" all'estremità 3', per un approccio sequenza-specifico.
La sonda TaqMan è un oligonucleotide complementare ad un tratto della sequenza amplificata dai primer. Usando una sofisticata tecnologia laser, con fibre ottiche dedicate per ogni singolo pozzetto, è possibile l'irradiazione e la rivelazione simultanea di fluorescenza dei campioni, emessa dai fluorofori VIC e FAM. La fluorescenza è rilevata da una telecamera CCD, il tutto sotto controllo di un computer.
Il rilevamento della quantità di OGM avviene mediante una quantificazione relativa che necessita la presenza di un controllo interno, lectina, che, amplificato, da' la possibilità, con un confronto di tipo relativo, di calcolare la percentuale di DNA transgenico usando una retta di riferimento generata con una serie scalare di DNA standard di soia transgenica da 0.1 a 5%; questo permette il calcolo della quantità iniziale di soia GM presente nei campioni in oggetto. Il metodo di calcolo è basato sul rapporto tra DNA transgenico e quantità di DNA totale, che permette di definire la percentuale in OGM all'interno del campione da testare.
Il limite di sensibilità dello strumento nell'analisi quantitativa su OGM è superiore allo 0.01%, questo limite corrisponde alla capacità di identificare equivalenti genomici inferiori alle 40 copie.
Nella tabella seguente è riportata la percentuale di soia geneticamente modificata contenuta nei campioni risultati positivi alla preliminare analisi qualitativa transgene-specifica.
| CAMPIONE | PERCENTUALE GMS |
| 1 | 0.18 % |
| 2 | 0.28% |
| 3 | 0.08% |
| 4 | 0.05% |
| 5 | 0.05% |
| 6 | 0.13% |
| 7 | 0.30% |
DISCUSSIONE
La considerevole crescita e diffusione delle biotecnologie ha investito numerosi settori produttivi tra cui il mondo dell'agricoltura. Sono numerose le specie vegetali geneticamente modificate e più di trenta hanno ricevuto il permesso di coltivazione e vendita in molte nazioni di tutto il mondo.
Nell'ambito della comunità europea, lo sviluppo della coltivazione di varietà geneticamente modificate è stato rigidamente regolamentato e la stessa commercializzazione di generi alimentari con ingredienti ottenuti da organismi geneticamente modificati è stata sottoposta a specifiche normative (14, 15) che impongono la precisa identificazione ed etichettatura del prodotto utilizzato.
La necessità di adeguarsi alle direttive legislative ha reso pertanto necessario la predisposizione di metodologie analitiche in grado di rilevare la presenza delle componenti transgeniche negli alimenti.
Per una precisa e accurata analisi qualitativa è indispensabile un efficiente metodo di estrazione.
Le bevande di soia vengono prodotte mediante trattamenti chimici e fisici, che possono modificare considerevolmente l'ammontare e la quantità del DNA dei vari ingredienti. La ripetibilità e la riproducibilità dei metodi di estrazione può quindi variare molto da campione a campione e quindi i protocolli sono stati adattati alla tipologia merceologica del campione.
La scelta e l'ottimizzazione delle procedure di estrazione del DNA, vengono effettuate in base alla capacità di eliminare i potenziali componenti inibitori, importante per la riuscita della PCR.
I cinque protocolli di estrazione, utilizzati in questo studio, presentano diversi approcci di estrazione e purificazione del DNA: la metodica Mag-easy e Wizard utilizzano particelle di silice magnetizzata per eliminare step multipli di centrifugazione e l'utilizzo di solventi organici. Inoltre a differenza dei sistemi basati su colonna, QIAamp DNA e DNeasy Plant, le particelle magnetiche legate agli acidi nucleici possono, essendo in soluzione, incrementare la loro cinetica ed efficienza.
Il metodo di estrazione suggerito dalla QIAGEN, ricorre invece all'uso di colonne, che necessitano di numerosi step di centrifugazione, sfruttando le proprietà di legame di membrane basate su un silica-gel.
Il metodo CTAB permette l'isolamento di grandi quantità di DNA, mediante precipitazione con solventi organici. La qualità e quantità dei DNA estratti mostrano, che il metodo CTAB è l'unico che ha permesso la visualizzazione e la quantificazione dei DNA di tutti i sette campioni.
Il protocollo di estrazione CTAB è risultato pertanto il più efficiente, seguito dal protocollo DNeasy Plant Maxi Kit, Mag-easy, QIAamp DNA Mini Kit ed infine Wizard.
La validità di un metodo di estrazione è condotta oltre che sull'efficienza, anche su altri parametri quali tempi di esecuzione e costi su base di routine.
Dalla valutazione complessiva di questi valori si può concludere che il metodo CTAB presenta i migliori requisiti come metodo di estrazione da bevande di soia, dimostrandosi il più efficace, efficiente, con tempo di estrazione medio lungo, svantaggio controbilanciato da bassi costi, dovuti alla preparazione di reagenti nell'ambito del laboratorio senza ricorrere a kit commerciali.
Ottenuti dei DNA amplificabili, la buona riuscita di un'analisi qualitativa dipende dalla strategia adottata e dalla specificità dei primers, come si è potuto verificare, con l'utilizzo dei primers 35S-cf3/35S-cr4 in sostituzione dei primers 35S-1/2.
La PCR fornisce un mezzo realizzabile per l'identificazione di alimenti contenenti OGM.
Per la rivelazione di piante geneticamente modificate sono usati diversi sistemi di PCR: quello di screening, non specifico per una particolare pianta e quelli specifici per un determinato OGM.
Il vantaggio di un test specifico è l'inequivocabile determinazione di una individuale variante, mostrando un grado di sensibilità lievemente più alto rispetto al sistema di screening.
Approssimativamente 10-100 molecole di DNA sono individuabili usando una determinazione di screening qualitativa, mostrando un valore relativo inferiore allo 0.1%. La sensibilità diminuisce però con l'incremento del grado di processazione del cibo, come per le bevande di soia, mostrando un range di determinazione compreso tra 0.1 e 1%.
Una determinazione qualitativa mediante PCR solitamente è sufficiente per fare una dichiarazione sulla presenza di costituenti GM. Comunque se è necessario andare oltre la presenza o assenza di OGM e determinare l'ammontare dell'organismo geneticamente modificato presente, si ricorre all'analisi quantitativa Real Time PCR, mediante ABI PRISM 7700 Sequence Detection System.
Il limite di sensibilità delle procedure quantitative è dello 0.01% variando con le condizioni del prodotto. Come nelle analisi qualitative, più è grande il grado di processazione, più si riduce la sensibilità della procedura.
Tutti i campioni di bevande di soia, risultati qualitativamente positivi alla presenza del transgene CTP/EPSPS, sono stati analizzati in PCR quantitativa, i dati ottenuti indicano una presenza del gene esogeno della soia inferiore all'1%.
Pertanto, le sette bevande di soia, tutte contenenti Soia Geneticamente Modificata (GMS), rientrerebbero nei limiti di legge e non necessiterebbero di certificazione, se non fosse per la presenza di due campioni provenienti da agricoltura biologica ed altri due destinati all'infanzia, per i quali la normativa vigente ( Decreto Ministeriale 4 Agosto 2000, G.U. n° 211 09/09/2000 e D.P.R. 7 Aprile 1999 n° 128) prevede un limite di tolleranza zero.
Questi risultati portano all'evidenza le conseguenze della notevole diffusione di soia GM e degli inevitabili problemi di segregazione dei prodotti OGM-free durante le fasi di stoccaggio e trasporto delle granaglie fino ai punti di destinazione; queste fasi sono quindi legate a possibile contaminazione crociata tra raccolti diversi.
BIBLIOGRAFIA
1. Malik, J., G. Barry and G. Kishore (1989) The herbicide glyphosate. BioFactors 2, 17-25
2. Padgette, S. R., Kolacz, K. H., Delanny, X., Re, D. B. et al. (1995) Development, Identification, and Characterization of a Glyphosate-Tolerant Soybean Line. Crop Science 35, 1451-1462
3. Padgette, S. R., G. della-Cioppa, D. M. Shah, R. T. Fraley, R. T. and G. M. Kishore (1989) Selective herbicide tolerance through protein engineering. P.441-476. In Schell et al. Cell culture and somatic cell genetics of plants. vol. 6 Academic Press. New York
4. Padgette, S. R., D. B. Re, G. F. Barry, D. E. Eichhltz, X. Delannay, R. L. Fuchs, G. M. Kishore, and R. T. Fraley (1995) New Weed Control Opportunities: Development Of glyphosate-tolerant soybeans. In S. O. Duke Herbicide resistant crops. CRC Press, Boca Raton, FL.
5. Shah, D., R. Horsch, R. H. Klee, G. Kishore, J. Winter, N. Turner, C. Hironaka, P. Sanders, C. Gasser, S. Aykent, N. Siegel, S. Rogers and R. Fraley (1986) Engineering herbicide tolerance in transgenic plants. Science 233, 478-481
6. Monsanto patent WO 92/04449, 132-136
7. della-Cioppa, G., Bauer, S. C., Klein, B. K., Shah, D. M., Fraley, R. T. und Kishore, G. (1986) Translocation of the precursor of 5-enol-pyruvyl- shikimate-3-phosphate synthase into chloroplasts of higher plants in vitro. Proc. Natl. Acad. Sci. USA. 83, 6873-6877
8. Steinrucken, H. C. und Amrhein, N. (1980) The herbicide glyphosate is a potent inhibitor of 5-enolpyruvyl shikimic acid-3-phosphate synthase. Biochem. Biophys. Res. Comm. 94, 1207-1212
9. Barry, G., Kishore, G., Padgette, S., Taylor, M., Kolacz, K., Weldon, M., Re, D., Eichholtz, D., et al. (1992) Inhibitors of amino acid biosynthesis: strategies for Imparting glyphosate tolerance to crop plants. p 139-145. In Singh, B. K. et al Biosynthesis and molecular regulation of amino acids in plants. Am. Soc. Plant Physiologists, Rockville, MD.
10. Ruff, T., D. Eichholtz, D. Re, S. Padgette, and G. Kishore (1991) Effects of amino acid substitution on glyphosate tolerance and activity of EPSPS Plant Physiol Suppl. 96, 94
11. Kay, R., A. Chan, M. Daly and J. McPherson (1987) Duplication of the CaMV 35S promoter sequences creates a strong enhancer for plant genes. Science 236, 1299-1302
12. Council Directive on the Deliberate Release into the Environment of Genetically Modified Organisms, 23 April 1990, EC Directive 90/220/EEC
13. Marcello Pierini (2000) Emissione deliberata di organismi geneticamente modificati disciplina e tutela del consumatore. Estratto dal Nuovo Diritto Agrario. Università degli studi di Urbino Carrefour Europeo
14. Regolamento Comunitario 49/2000 della Commissione 10/1/2000 G.U.CE 11/1/2000
15. Regolamento Comunitario 50/2000 della Commissione 10/1/2000 G.U.CE 11/1/2000
16. .Meyer R., Chardonnens F., Hubner P. and Luthy J. (1996) Polymerase chain reaction (PCR) in the quality and safety assurance of food. Detection of soya in processed meat products. Z Lebensm. Unters. Forsch. 203, 339-344
17. Niederhauser, Ch., Gilgen, M., and Meyer, R., (1996) Genetically engineered food plants: research and detection with DNA-analytical methods. Mitt. Gebiete Lebensm. Hyg. 87, 307-367
18. G. Van den Eede, M. Lipp, F. Eyquem, E. Anklam (2000) Validation of an Analytical Method for the Detection of GMO-derived DNA in Processed Foodstuffs. European Commission, DG Joint Research Centre
19. Hagen M., Beneke B. (2000) Detection of genetically modified soy (Roundup-Ready) in processed food products Berl Munch Tierarztl Wochenschr 113, 454-458
20. Hemmer, W., (1997) Foods derived from genetically modified organisms and detection methods. BATS Report 2/97
21. Koppel, E., Stadler, M., Luthy, J. and Hubner, P. (1997) Sensitive Method for the detection of the genetically engineered soybean "Roundup Ready". Mitt. Gebiete Lebensm. Hyg. 88, 164-175
22. Rolf Meyer and Etienne Jaccaud (1997) Detection of genetically modified soya in processed food products: development and validation of a PCR assay for the specific detection of Glyphosate-Tolerant Soybeans Proceedings of the Ninth European Conference on Food Chemistry (EURO FOOD CHEM IX) Event No. 220, Vol. 1, p. 23-28. (Interlaken, Switzerland, September 24-26
23. Andreas Zimmermann, Jurg Luthy, Urs Pauli (1998) Quantitative and qualitative evaluation of nine different extraction methods for nucleic acids on soya bean food samples Z Lebensm Unters Forsch A 207, 81-90
24. Carolyn D. Hurst, Angus Knight and Ian J. Bruce (1999) PCR detection of genetically modified soya and maize in foodstuffs Molecular Breeding 0, 1-8
25. Pietsch K., Waiblingher U., Brodman P., and Wurz A. (1997) Screeningverfahren zur Identifizierung gentechnisch veranderter Lebensmittel. Deutsche Lebensmittel-Rundschau Heft 2, 35-39
26. Vodkin L. O., Rhodes P. R., Goldherg R. B. (1983) cA lectin gene insertion has the structural features of a transposable element. Cell 34, 1023-1031
27. Screening method for the identification of genetically modified organisms (GMO) in food. Detection of the CaMV 35S promoter and NOS terminator by means of polymerase chain reaction (PCR). DG JRC. Environment Institute, Consumer Protection and Food Unit
28. Anklam, E. (1998) The JRC validates a qualitative screening method for the detection of genetically modified soy beans and maize in food raw material. Press release. JRC, T. P. 740. I-21020 Ispra
29. The analysis of Food Samples for the Presence of Genetically Modified Organisms (2000) EUROPEAN COMMISSION Joint Research Centre, Ispra
30. Lipp M., Brodmann P., Pietsch K., Pauwels J., Anklam E., Meyer R., Muller W., Wurtz A. et al. (1999) IUPAC collaborative trial study of a method to detect genetically modified soy beans and maize in dried powder. J. AOAC 82, 923-928
31. L. Grohmann, R. Stasche and F. R. Lauter (2000) Real-Time PCR as a Quantitative Method to Detect Genetic Modifications in Food Products Munich, Germany, AOACI Europe/GDCh/DVG
32. .Higuchi, R., Fockler, C., Dollinger, G., and Watson, R. (1993) Kinetic PCR: Real time monitoring of DNA amplification reactions. Biotechnology 11, 1026-1030
33. Livak, K. J., Flood, S.J. A., Marmaro, J., Giusti, W., and Deetz, K. (1995) Oligonucleotides With fluorescent dyes at opposite ends provide a quenched probe system useful for detecting PCR product and nucleic acid hybridization. PCR Methods and Applications 4, 357-362
